Resumo
O diabetes relacionado à infecção pode surgir como resultado da destruição de células β associada a vírus. Os dados clínicos sugerem que a síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2), causando a doença coronavírus 2019 (COVID-19), prejudica a homeostase da glicose, mas faltam evidências experimentais de que o SARS-CoV-2 pode infectar o tecido pancreático . No presente estudo, mostramos que o SARS-CoV-2 infecta células do pâncreas exócrino e endócrino humano ex vivo e in vivo. Demonstramos que as células β humanas expressam proteínas de entrada viral e o SARS-CoV-2 infecta e se replica em ilhotas humanas cultivadas. A infecção está associada a alterações morfológicas, transcricionais e funcionais, incluindo números reduzidos de grânulos secretores de insulina em células β e secreção de insulina estimulada por glicose prejudicada. Em exames pós-morte de corpo inteiro COVID-19, detectamos a proteína do nucleocapsídeo SARS-CoV-2 em células exócrinas pancreáticas e em células que se coram positivamente para o marcador de células β NKX6.1 e estão nas proximidades das ilhotas de Langerhans em todos os quatro pacientes investigados. Nossos dados identificam o pâncreas humano como um alvo da infecção por SARS-CoV-2 e sugerem que a infecção de células β pode contribuir para a desregulação metabólica observada em pacientes com COVID-19.
A Principal
Inicialmente, a pandemia de COVID-19, causada pelo SARS-CoV-2, foi considerada uma doença pulmonar exclusiva, podendo levar a graves sintomas respiratórios 1 . Nesse ínterim, os estudos experimentais e clínicos acumulados sugeriram que o SARS-CoV-2 também pode causar lesões nos rins, coração, cérebro e órgãos gastrointestinais e endócrinos 2 , 3 , 4 , 5 , 6 , 7 . O tropismo da SARS-CoV-2 para tecidos distintos é governado por fatores celulares expressos em células-alvo, como o receptor de entrada viral da enzima conversora de angiotensina 2 (ACE2) 8 e a serina protease 2 transmembrana (TMPRSS2) 8 . ACE2A expressão do RNA mensageiro 9 , 10 , 11 , 12 , 13 e da proteína 12 , 13 , 14 nas ilhotas de Langerhans foi relatada, mas ainda não foi mostrada, para permitir a entrada de SARS-CoV-2 9 , 12 , 15 . Diabetes mellitus apresenta Janus como em COVID-19 (refs. 3 , 16 ): primeiro, diabetes pré-existente é uma comorbidade altamente prevalente observada em 11–22% dos pacientes e, como tal, aumenta o risco de uma doença grave, exigindo mais intensidade intervenções e aumento da mortalidade 17 , 18 ,19 , 20 , 21 , 22 . Em segundo lugar, a infecção por SARS-CoV-2 parece afetar o pâncreas exócrino, manifestando-se como pancreatite em 32,5% dos pacientes gravemente enfermos 23 , e aumento do pâncreas e níveis anormais de amilase ou lipase em 7,5-17% dos pacientes 9 , 22 . Terceiro, desregulação metabólica foi observada em pacientes com COVID-19 como: (1) aumento da hiperglicemia em pacientes com diabetes tipo 2 24 ; (2) cetoacidose em 2–6,4% dos pacientes diabéticos e não diabéticos 18 , 25; e (3), em estudos de caso relatando cetoacidose na infecção por SARS-CoV-2, acompanhada por (4) diabetes mellitus tipo 1 de início recente (DM1) na ausência de autoanticorpos 26 , 27 , 28 . Em um estudo de coorte de pacientes com diabetes, hiperglicemia foi relatada em mais de 50% de todos os casos, e quase um terço apresentou cetoacidose diabética 29 . Por fim, um estudo multicêntrico encontrou um aumento de 80% no DM1 de início recente em crianças durante a pandemia de COVID-19 30 . De acordo, uma meta-análise recente resume que COVID-19 grave está associado a níveis elevados de glicose no sangue 31. No entanto, a prova formal de SARS-CoV-2 como um vírus com trópico de células β, potencialmente levando ao diabetes, ainda está faltando, e a única evidência correlativa está à luz de achados experimentais e clínicos conflitantes 13 , 22 , 32 , 33 , 34 . Resultados agudos e de longo prazo coletados prospectivamente em casos de diabetes de início recente, juntamente com interpretações ponderadas de dados emergentes até o esclarecimento final desse debate, são necessários 33 .
Consequentemente, não está claro se e como o SARS-CoV-2 pode desencadear lesão de células β, mas pode ocorrer por meio de ablação de células β imunomediada ou perturbação direta da função de células β, ambos eventualmente levando à chamada infecção diabetes relacionado à atual classificação da Organização Mundial da Saúde (OMS) 35 . Evidências recentes sugerem que o SARS-CoV-2 pode infectar células endócrinas humanas in vitro 12 , 36 . No entanto, esta descoberta foi obtida com células-tronco humanas imaturas derivadas de células-tronco que expressam ACE2 e TMPRSS2, e a replicação viral ou impacto na função das células-β não foi analisada em detalhes 36. A expressão de ACE2 ou TMPRSS2 também foi detectada em células exócrinas e endócrinas em tecido pancreático humano; no entanto, vários padrões de expressão em diferentes tipos de células pancreáticas foram relatados 9 , 10 , 12 , 13 , 14 , 15 , 34 , 36 , 37 , 38 . Assim, é imperativo esclarecer se as células endócrinas pancreáticas humanas organizadas dentro de uma ilhota de Langerhans são permissivas e afetadas pela infecção por SARS-CoV-2, e elucidar os mecanismos subjacentes a uma disfunção endócrina potencial associada a COVID-19 (refs. 16 , 21 , 22, 36 ).
No presente estudo, nós (1) definimos os padrões de expressão de ACE2 e TMPRSS2 em tipos de células endócrinas e exócrinas pancreáticas humanas, (2) empregamos culturas de ilhotas pancreáticas humanas para demonstrar a suscetibilidade à infecção por SARS-CoV-2 e replicação viral em células β e (3) mostraram que a infecção por SARS-CoV-2 afeta a secreção de insulina estimulada por glicose (GSIS). Além disso, (4) visualizamos as partículas virais se replicando nas células pancreáticas endócrinas e definimos seus padrões de localização subcelular e, finalmente (5) apresentamos exemplos de padrões de infecção de múltiplos órgãos, incluindo o pâncreas de pacientes que morreram de COVID-19.
Resultados
Expressão de ACE2 e TMPRSS2 em células endócrinas e uma subpopulação ductal
Como a expressão de ACE2 e TMPRSS2 pancreática está atualmente em debate 12 , 13 , 34 , 37 , iniciamos nossa análise de validação com dois anticorpos de referência (ab15348 e ab92323, Abcam; Extended Data Fig. 1 ), que foram anteriormente extensivamente caracterizados em imunofluorescência e estudos de imunohistoquímica (ACE2 (refs. 12 , 36 , 39 , 40 , 41 , 42 ) e TMPRSS2 (refs. 43 , 44 , 45 , 46 , 47)). Primeiro, a análise quantitativa de PCR (qPCR) de ACE2 e TMPRSS2 em células Calu-3 de pulmão humano e células EndoC-βH1, um modelo de células β humanas 48 , foi realizada e revelou níveis detectáveis de RNA de ambas as proteínas de entrada viral (Figura de dados estendidos . 2a ). Imunotransferência confirmaram detecção da massa molecular correcto da ~ 110/120 kDa e recentemente relatado 12 , 49 curto 50-kDa isoformas de ACE2 endógeno em ambos os tipos celulares (dados estendidos Fig. 2b ). Descobrimos que todas as isoformas de ACE2 são expressas em tecido pancreático humano recém-congelado compreendendo tipos de células exócrinas e endócrinas (Dados estendidos Fig. 2b) Da mesma forma, o anticorpo TMPRSS2 detectou proteínas de 54 kDa e 26 kDa, consistentes com formas glicosiladas de TMPRSS2 de comprimento total e o domínio de serina protease clivado, conforme relatado anteriormente 50 (Dados estendidos Fig. 2c ). Notavelmente, a expressão de ACE2 e TMPRSS2 das diferentes isoformas variou entre os participantes (dados estendidos Fig. 2b, c ), em linha com os achados anteriores 12 . A imagem de imunofluorescência revelou padrões de coloração semelhantes para a expressão de ACE2 ou TMPRSS2 em culturas de interface ar-líquido diferenciadas de células epiteliais das vias aéreas humanas primárias (HAECs) 51 , bem como em células EndoC-βH1 (Dados estendidos Fig. 2d, e) A pré-incubação do anticorpo anti-ACE2 empregado com um peptídeo bloqueador de correspondência de epítopo anulou a detecção de ACE2 em células EndoC-βH1 (Dados Estendidos Fig. 2e ).
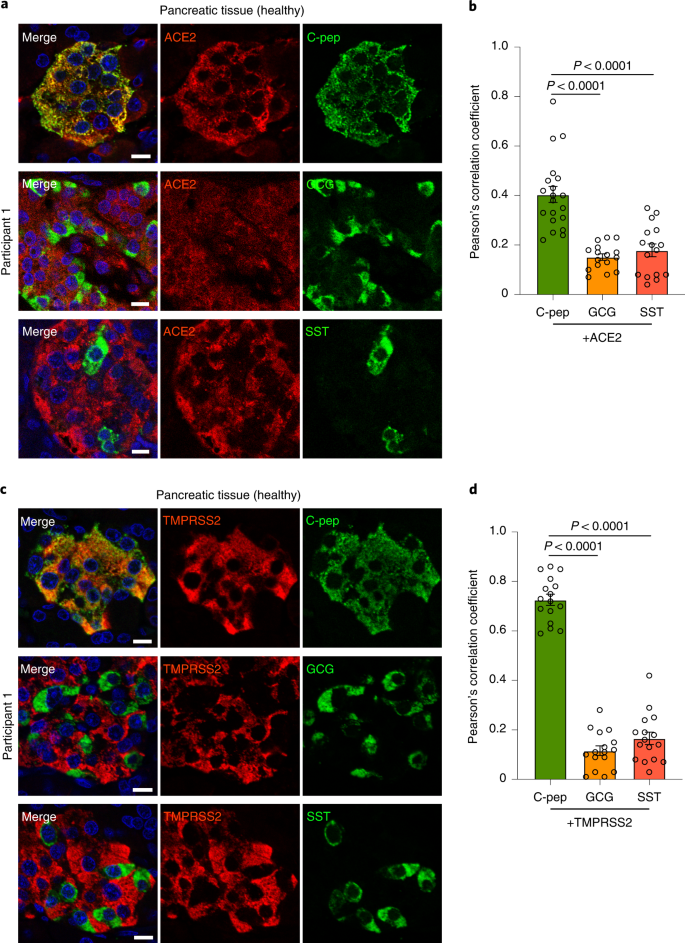
Após a validação da especificidade do anticorpo, visualizamos a expressão de ACE2 e TMPRSS2 em seções de tecido derivadas de cinco pancreatos humanos histologicamente saudáveis. A coloração por fluorescência de ambos os fatores de entrada do SARS-CoV-2 foi observada nas ilhotas de Langerhans em todas as amostras (Fig. 1a, c , e Dados Estendidos nas Figs. 3 e 4 ). Expressão forte de ACE2 foi detectada em células endoteliais (dados estendidos Fig. 3a-c , pontas de setas brancas) e em uma subpopulação de células ductais positivas para citoqueratina 19 (CK19) (dados estendidos Fig. 3d ). Sinais moderados de ACE2 foram observados em células endócrinas (Fig. 1a e Dados Estendidos Fig. 3a – c) e a detecção foi evitada pelo peptídeo de bloqueio de ACE2 durante a imunocoloração (Dados Estendidos Fig. 3e ). ACE2 foi apenas fracamente expresso em células acinares positivas para a proteína 4 de ligação a GATA (dados estendidos Fig. 3f ). Da mesma forma, TMPRSS2 foi detectado no compartimento endócrino (Fig. 1c e Dados Estendidos Fig. 4a-c ) e em alguns dutos (Dados Estendidos Fig. 4d ). A expressão de TMPRSS2 em células acinares quase não foi detectável (Dados estendidos Fig. 4e ). A co-coloração para tipos de células endócrinas e proteínas de entrada viral revelou um padrão de coloração heterogêneo entre os cinco doadores com coeficientes variáveis (Fig. 1b, d) Os coeficientes mais altos foram encontrados para células β-peptídeo C (C-pep) co-coradas para ACE2 (média: 0,40 (mínimo – máximo: 0,22–0,78)) e TMPRSS2 (média: 0,73 (mínimo – máximo: 0,59 –0,86)) (Fig. 1b, d ). As células α e δ que expressam glucagon (GCG) ou somatostatina (SST), respectivamente, revelaram um ACE2 menor (média: 0,15 (mínimo – máximo: 0,07–0,23); média: 0,18 (mínimo – máximo: 0,04–0,35 )) ou TMPRSS2 (média: 0,12 (mínimo – máximo: 0,01–0,28); média: 0,17 (mínimo – máximo: 0,03–0,42)) fração duplo-positiva com menos variância entre os cinco pacientes (Fig. 1b, d ). Assim, as células pancreáticas exócrinas e endócrinas expressam fatores de entrada da SARS-CoV-2.
a , c , Seções de tecido pancreático adulto de cinco participantes saudáveis, coradas com anticorpos contra ACE2 ( a ; vermelho) ou TMPRSS2 ( c ; vermelho) e C-pep (verde), GCG (verde) ou SST (verde). Os núcleos das células foram visualizados usando DAPI (azul). Seções confocais representativas do participante 1 são mostradas; para os participantes 2–5, consulte as Figs de dados estendidos. 3 e 4 . Barras de escala, 10 µm. b , d , Colocalização de marcadores específicos de células C-pep, GCG ou SST com ACE2 ( b ) ou TMPRSS2 ( d ) foi analisada usando coeficientes de correlação de Pearson e Fiji. Os números de pancreatos humanos saudáveis investigados foram os seguintes: ACE2 / C-pep:n = 5; TMPRSS2 / C-pep: n = 4; ACE2 / SST ou GCG: n = 4; TMPRSS2 / SST ou GCG: n = 4. Quatro ilhotas diferentes de Langerhans foram analisadas por paciente. Os dados são apresentados como média ± sem quantificados para cada participante e combinação de coloração. Foi utilizada a análise de variância (ANOVA) comum com pós-teste de Tukey.
SARS-CoV-2 replica em ilhotas pancreáticas humanas
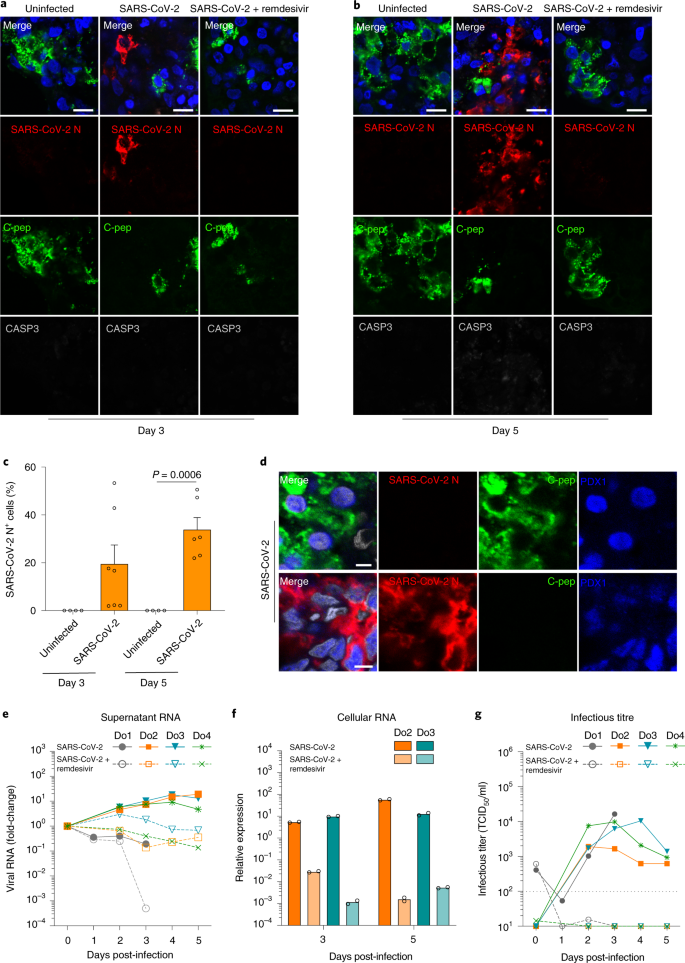
Para determinar a suscetibilidade à infecção ex vivo, ilhotas pancreáticas humanas isoladas de quatro doadores humanos foram expostas ao SARS-CoV-2, e a expressão do pico viral (S) e da proteína do nucleocapsídeo (N), bem como marcadores de células endócrinas, foi analisada . As proteínas S e N não foram detectadas no dia 1 (não mostrado), mas tornaram-se prontamente detectáveis nos dias 3 (Fig. 2a ) e 5 (Fig. 2b ) pós-infecção (Dados estendidos Fig. 5 ). Ilhotas pancreáticas tratadas com remdesivir 5 µM, um inibidor da polimerase com potente atividade anti-SARS-CoV-2 in vitro 52, não corou positivo para as proteínas S ou N, indicando supressão da replicação do SARS-CoV-2. A quantificação da expressão da proteína N viral em ilhotas infectadas confirmou a infecção robusta variando entre 20% de células N-positivas no dia 3 e 34% no dia 5 por ilhota infectada (Fig. 2c ). Apenas algumas células coraram positivamente para caspase 3 clivada (CASP3) em todas as condições, sugerindo que não há aumento da apoptose nesta fase (Fig. 2a, b ). Embora algumas células exibissem dupla positividade para os hormônios pancreáticos C-pep / cromogranina A e as proteínas N / S virais, o que não foi observado para GCG e SST, a maioria das células infectadas com SARS-CoV-2 parecia não ter expressão de hormônio ( Fig. 2a, b, d e Dados Estendidos Fig. 5) A coloração com marcadores não endócrinos, como o marcador endotelial, molécula de adesão de células endoteliais de plaquetas (PECAM-1, CD31), o marcador ductal CK19 ou o marcador acinar quimiotripsina revelou apenas células positivas dispersas em preparações de ilhotas pancreáticas, tornando assim o viés dos resultados devido a impurezas de preparação improváveis (dados estendidos Fig. 6 ). Para sondar a identidade da linhagem de células infectadas, coramos para homoeobox 1 pancreático e duodenal (PDX1) e NKX6.1, ambos marcadores marcando principalmente células endócrinas no pâncreas adulto 53 , 54 , 55 , e descobrimos que as células hormonais negativas infectadas ainda eram positivo para PDX1 ou NKX6.1, sugerindo que são células endócrinas que perdem hormônios após a infecção (Fig. 2d) A quantificação de células N / NKX6.1 duplamente positivas por ilhota infectada do doador 3 no dia 5 após a infecção revelou que aproximadamente 21% das células β putativas estavam infectadas (Dados estendidos Fig. 6d, e ). Uma quantificação mais definida foi impedida pela quantidade de material analisável e pelo fato de que as células N / S-positivas da linhagem endócrina parecem ser insulino-negativas na infecção. No entanto, o aumento dos níveis intra e extracelulares de RNA viral das ilhotas na ausência de remdesivir indica replicação viral progressiva (Fig. 2e, f ). A replicação viral produtiva nas ilhotas de todos os doadores foi confirmada pelo aumento dos títulos virais infecciosos nos respectivos sobrenadantes (Fig. 2g) No tratamento com remdesivir, quase nenhum vírus infeccioso foi detectado nos sobrenadantes das ilhotas (Fig. 2g ), indicando inibição eficiente. Isso está de acordo com os baixos níveis de RNA viral (Fig. 2e, f ) e a ausência de proteína N ou S em análises de microscopia confocal (Fig. 2a, be Dados Estendidos Fig. 5 ) na presença de remdesivir. Assim, as ilhotas pancreáticas são suscetíveis à infecção por SARS-CoV-2, que pode ser inibida pelo remdesivir.
Ilhotas pancreáticas humanas foram falsamente tratadas com meio ou infectadas com SARS-CoV-2 e cultivadas na presença ou ausência de remdesivir (5 µM). As imagens exibidas representam três doadores. a , b , ilhotas do doador 2 fixadas 3 d ( a ) ou 5 d ( b ) pós-infecção foram coradas para proteína SARS-CoV-2 N (SARS-CoV-2 N, vermelho), C-pep (verde) , CASP3 clivado (branco) e núcleos (DAPI, azul). Imagens representativas são mostradas. Barras de escala, 10 µm. c , Quantificação da fração de células SARS-CoV-2 N-proteína-positivas em ilhotas infectadas de doadores 2 e 3 (ver dados estendidos Fig. 5para colorações de doador 3). Os dados são apresentados como média ± sem de sete (dia 3) e seis (dia 5) ilhotas infectadas individuais e de quatro ilhotas não infectadas (não pareado, bilateral, teste t de Student ). d , ilhotas do doador 3 foram infectadas e células infectadas com SARS-CoV-2 (SARS-CoV-2 N, vermelho), células β (C-pep, verde) e células de linhagem endócrina (PDX1, azul) foram visualizadas. Os núcleos corados com DAPI são pseudo-coloridos em branco. Barra de escala, 5 µm. As células infectadas com SARS-CoV-2 freqüentemente se tornaram negativas para o hormônio (C-pep), mas permaneceram com linhagem positiva (PDX1). As células não infectadas permaneceram duplamente positivas para C-pep e PDX1. e , os sobrenadantes das ilhotas (doadores 1-4) foram colhidos ao longo de 5 dias e SARS-CoV-2 ORF1b-nsp14foi quantificado por qPCR. Os dados são apresentados como os valores médios de duas repetições. Para cada doador (Do), as alterações em relação ao dia 0 (RNA de entrada viral) são visualizadas. f , o SARS-CoV-2 N foi quantificado em isolados celulares de RNA de doadores 2 e 3 nos dias 3 e 5 pós-infecção e normalizado para RNA GAPDH . Os dados são apresentados como valores médios de duas repetições. g , os sobrenadantes de todos os quatro dadores de e foram avaliados quanto à infecciosidade através de TCID 50 de titulação de ponto final. Os dados são apresentados como os valores médios de duas repetições e a linha pontilhada indica o limite inferior de quantificação.
Células endócrinas infectadas com SARS-CoV-2 mostram alterações subcelulares e funcionais
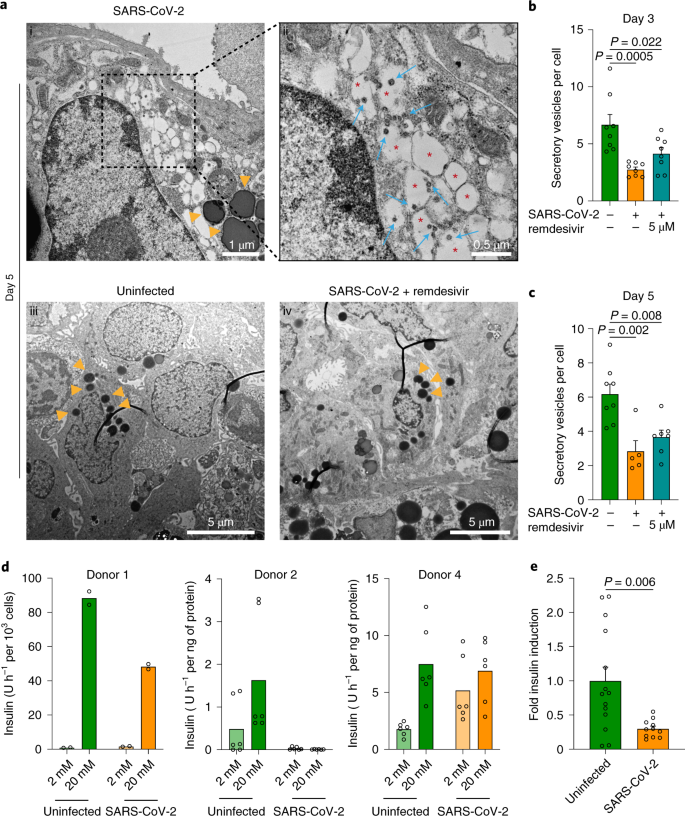
A infecção de células endócrinas por SARS-CoV-2 foi posteriormente analisada usando microscopia eletrônica de transmissão (TEM). Ilhotas pancreáticas de doadores de órgãos humanos 2 e 3 foram infectadas com SARS-CoV-2 e analisadas após 3 e 5 dias de cultura ex vivo (Fig. 3a e Dados estendidos Fig. 7 ). A infecção das células das ilhotas com SARS-CoV-2 resultou na dilatação e vacuolização do retículo endoplasmático (RE) – complexo do aparelho de Golgi, um achado sugestivo de estresse RE e edema corporal de Golgi 56 , 57 , 58 . Esses vacúolos continham partículas virais mostrando a morfologia do coronavírus 56 , 57 , 58, indicando células endócrinas produtivamente infectadas. As vesículas contendo virion foram formadas na região perinuclear e processadas para a superfície da célula. Além disso, a infecção ocorreu em células contendo vesículas secretoras que pareciam estar aumentadas e mantidas na região perinuclear (Fig. 3a ). O número de grânulos por célula em ilhotas infectadas diminuiu 2,2 vezes no dia 3 (Fig. 3b ) e 2,4 vezes no dia 5 (Fig. 3c ) após a infecção. Em contraste, não detectamos partículas virais intracelulares e observamos menos alterações morfológicas nas células das ilhotas tratadas com remdesivir (Fig. 3a e Dados Estendidos Fig. 7a) Assim, o SARS-CoV-2 se comportou em células endócrinas humanas semelhantes aos fenótipos TEM relatados anteriormente de células infectadas derivadas de pulmão e intestino 56 , 57 , 58 . Para analisar se a infecção por SARS-CoV-2 e as alterações subcelulares associadas das ilhotas afetam a função, avaliamos a resposta das ilhotas a um pulso alto de glicose. Descobrimos que GSIS foi induzido em todas as condições, mas a magnitude da indução de glicose baixa para alta foi reduzida em ilhotas infectadas (Fig. 3d, e) Digno de nota, a responsividade geral à glicose foi menor em preparações de duas ilhotas e variou entre as preparações, muito provavelmente devido às limitações da cultura ex vivo prolongada. Esses dados corroboram que o SARS-CoV-2 se replica nas células endócrinas e sugere que a infecção pode afetar a secreção de insulina dependente da glicose nas ilhotas pancreáticas. No entanto, para obter conclusões mais definitivas, serão necessários mais preparativos de ilhotas.
Ilhotas pancreáticas humanas do doador 3 foram infectadas com SARS-CoV-2 e cultivadas com ou sem remdesivir 5 µM, ou não foram infectadas. a , No dia 5, as ilhotas foram fixadas e seccionadas para análise TEM. A micrografia eletrônica (i) e a entrada ampliada (ii) da preparação infectada mostram células com vesículas secretoras endócrinas (pontas de seta laranja) e vacúolos de Golgi dilatados (asteriscos vermelhos) contendo partículas de vírus (setas azuis). Vacúolos e partículas virais estavam ausentes nas amostras não infectadas (iii) e tratadas com remdesivir (5 µM) (iv) (ver dados estendidos Fig. 7a para outras micrografias com ampliações iguais como em i e ii). b , c , vesículas secretoras endócrinas do doador 3 foram identificadas manualmente por dois indivíduos independentes no dia 3 (b ) ou 5 ( c ) pós-infecção e quantificado às cegas usando Fiji. Os dados são apresentados como média ± sem de 8 imagens TEM (dia 3) e 8, 5 e 7 imagens TEM para ilhotas não infectadas, infectadas e tratadas com remdesivir (dia 5), respectivamente, contendo em média 19 núcleos. A significância estatística foi calculada por ANOVA de um fator comum com o pós-teste de Tukey. d , e , No dia 3 pós-infecção, a funcionalidade das ilhotas dos doadores 1, 2 e 4 foi analisada por GSIS estático. As ilhotas foram expostas primeiro a 2 mM e depois a 20 mM de glicose durante 1 h cada, e a secreção de insulina no tampão foi determinada por ELISA de insulina ultrassensível. A secreção de insulina calculada indicada nas barras correspondentes ( d ) e calculada a média como induções de dobra ( e) é mostrado. Os dados são apresentados como média ± sem de duas repetições técnicas (doador 1) e seis repetições (doadores 2 e 4). A significância estatística da indução de insulina dobrada foi calculada usando um teste t de Student de dois lados não pareado .
Alterações transcricionais em ilhotas humanas após infecção por SARS-CoV-2
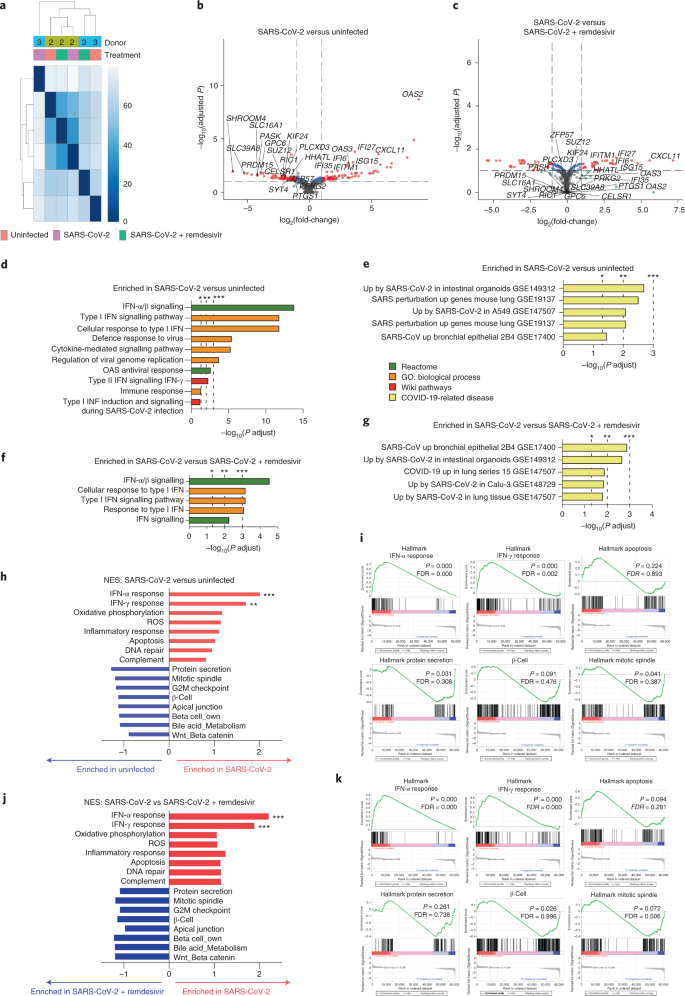
Para avaliar as alterações transcricionais potenciais induzidas por SARS-CoV-2, realizamos sequenciamento de RNA em massa (Smart-Seq2) de ilhotas humanas cultivadas não infectadas e infectadas (com ou sem remdesivir) de dois doadores em uma análise exploratória. Primeiro, os respectivos transcriptomas obtidos 5 dias após a infecção foram agrupados. A maior parte da variância da amostra foi determinada pelas duas preparações de ilhotas diferindo também no sexo do doador (Fig. 4a ). No entanto, os transcriptomas de células infectadas com SARS-CoV-2 separaram-se claramente das contrapartes não infectadas, enquanto o tratamento com remdesivir resultou em agrupamento intermediário (Fig. 4a ). Entre os principais genes regulados positivamente em ilhotas infectadas com SARS-CoV-2 estavam vários genes estimulados por interferon (IFN) (ISGs), como IFITMs 59, OAS2, IFI27 e ISG15, enquanto os genes ligados à fisiologia das células β ou diabetes 59 , 60 , 61 , 62 , 63 , 64 , 65 , como SYT4, PASK, PEX6 e PLCXD3, foram significativamente regulados para baixo (Fig. 4b ). Digno de nota, os ISGs foram regulados positivamente não apenas após a infecção por SARS-CoV-2, em comparação com os casos não infectados, mas também em ilhotas tratadas com remdesivir (Fig. 4c) A análise do termo da ontologia gênica (GO) confirmou o início de uma reação de defesa celular transcricional em resposta à infecção por SARS-CoV-2. Termos como ‘resposta de defesa ao vírus’ e ‘regulação da replicação do genoma viral’ foram fortemente regulados positivamente após a infecção por SARS-CoV-2 (Fig. 4d ). A comparação da infecção por SARS-CoV-2 com culturas infectadas tratadas com remdesivir revelou um enriquecimento semelhante, mas menos pronunciado, de termos relacionados a INF, como ‘sinalização de IFN α / β’ e ‘via de sinalização de IFN tipo I’, indicando uma reversão parcial das alterações transcricionais (Fig. 4f ). Um padrão semelhante foi observado ao focar em termos de doença relacionados a COVID-19 (Fig. 4e, g) A análise de enriquecimento do conjunto de genes (GSEA) confirmou ainda o enriquecimento da sinalização de IFN em ilhotas infectadas com SARS-CoV-2 contra ilhotas infectadas não infectadas e tratadas com remdesivir (Fig. 4h-k ). Além disso, uma tendência indicando perda de identidade de células β, conforme revelado por vários conjuntos de genes 66 , bem como defeitos na secreção de proteínas em ilhotas infectadas por vírus, pode ser detectada (Fig. 4h-k ). Vice-versa, esses defeitos foram atenuados no tratamento com remdesivir, indicando que as alterações observadas são causadas pela infecção por SARS-CoV-2, o que está de acordo com nossos experimentos funcionais ex vivo (Fig. 3) Assim, em um nível transcricional, as ilhotas infectadas mostram reações de defesa inatas e alterações transcricionais indicativas de perda de identidade das células β. Digno de nota, a infecção viral e a resposta ao IFN tipo I demonstraram desencadear o desenvolvimento de DM1 em indivíduos com predisposição genética 67 , 68 , 69 . No entanto, mais estudos com amostras maiores serão necessários para confirmar essas conclusões.
Ilhotas pancreáticas humanas de doadores 2 e 3 foram infectadas com SARS-CoV-2 e cultivadas com ou sem remdesivir 5 µM, ou não infectadas, e preparadas para RNA-seq. a , mapa de calor de expressão Smart-seq2 que ilustra a proximidade entre diferentes condições de tratamento e experimentos ( n = 2 doadores de ilhotas). b , c , Gráficos de vulcão com genes representados de interesse para comparação de ilhotas infectadas com SARS-CoV-2 e ilhotas não infectadas ( b ) ou tratadas com remdesivir ( c ). Genes significativos foram destacados em azul ( P ajustado <0,1) e genes significativos com um log 2 (mudança de dobra)> | 1 | em vermelho. A significância estatística foi testada usando DESeq2 (ref.104 ). d , f , Seleção de conjuntos de genes significativamente enriquecidos comparando genes diferencialmente expressos em ilhotas infectadas com SARS-CoV-2 versus ilhotas não infectadas ( d ) ou versus ilhotas tratadas com remdesivir ( f ) em análises de super-representação em bancos de dados comuns; o teste de enriquecimento para significância foi realizado usando g: Profiler 105 . e , g , Seleção de conjuntos de genes significativamente enriquecidos em análises de super-representação contra termos de doenças relacionadas a COVID-19 comparando genes diferencialmente expressos em ilhotas infectadas com SARS-CoV-2 versus não infectadas ( e ) ou versus tratadas com remdesivir ( g) ilhotas; o teste de enriquecimento para significância foi realizado usando EnrichR 106 (*** ajustado P <0,001, ** ajustado P <0,01, * ajustado P <0,05). h , j , Pontuações de enriquecimento normalizado para uma seleção de conjuntos de genes em GSEA de ilhotas infectadas com SARS-CoV-2 em comparação com ilhotas não infectadas ( h ) ou tratadas com remdesivir ( j ) (FDR; *** P <0,001, * * P <0,01, * P <0,05). ROS, espécies reativas de oxigênio. i , k , gráficos de GSEA para conjuntos de genes selecionados de ilhotas infectadas com SARS-CoV-2 em comparação com não infectados ( i) ou para ilhotas tratadas com remdesivir ( k ). A taxa de descoberta falsa (FDR) foi determinada usando a ferramenta de desktop GSEA (Broad Institute) 107 conforme detalhado em Métodos .
Infecção cruzada de órgãos e pancreática durante COVID-19 grave
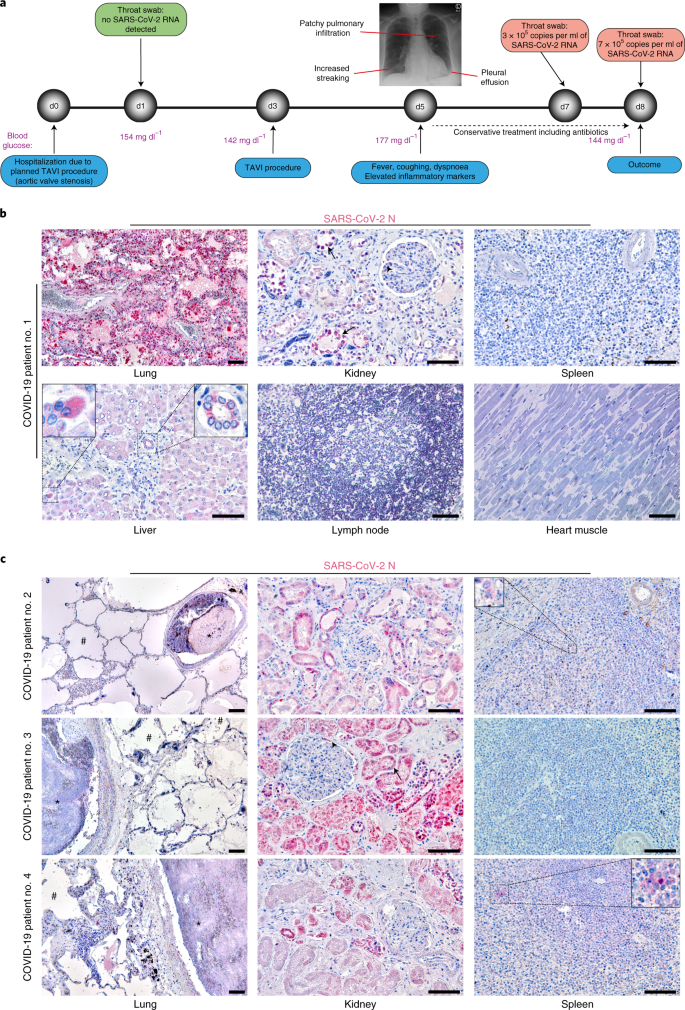
Para examinar se a infecção observada ex vivo de ilhotas também ocorre naturalmente no tecido pancreático, realizamos estudos post mortem em quatro indivíduos que morreram de COVID-19 (Tabela 1 ). Um paciente era SARS-CoV-2 negativo na admissão ao hospital, mas desenvolveu sintomas respiratórios 4 dias depois, e testes consecutivos de SARS-CoV-2 revelaram níveis crescentes de RNA viral em esfregaços nasofaríngeos analisados (Fig. 5a ). A história médica pregressa incluía diabetes tipo 2 controlado com metformina, hipertensão arterial e doença arterial coronariana (Tabela 1 ). Sob terapia conservadora, o paciente piorou rapidamente, acompanhado por uma demanda progressiva de oxigênio, e morreu devido a insuficiência respiratória 3 dias após o início dos sintomas clínicos (Fig. 5a) Os outros três pacientes apresentaram curso de doença mais longo, variando de 9 a 28 dias de internação e 6 a 24 dias de ventilação antes do óbito (Tabela 1 ). Um paciente era diabético em uso de medicação oral e um paciente apresentava insuficiência pancreática exócrina. Esses três pacientes desenvolveram lesão renal aguda em vários estágios. No total, dois pacientes morreram de insuficiência respiratória, um de sangramento gastrointestinal e um de insuficiência multiorgânica (Tabela 1 ).
a , Linha do tempo da evolução da doença do paciente 1, hospitalizado devido a um procedimento planejado de implante de válvula aórtica transcateter (TAVI), incluindo níveis de glicose no sangue durante a hospitalização e raio-X torácico no dia 5 de hospitalização. b, Vários órgãos do paciente 1 foram corados para proteína N viral (vermelho). Secções do pulmão mostraram forte coloração de macrófagos alveolares eliminados, bem como de células de revestimento alveolar. No rim, as células epiteliais do túbulo (setas) são positivas para a proteína N, assim como algumas células dentro do glomérulo (ponta da seta). O baço não mostra células positivas. No fígado, a proteína N é detectada em algumas células do ducto biliar como hepatócitos bem isolados, enquanto os linfonodos e o músculo cardíaco parecem negativos. Imagens representativas de quatro seções de biópsia do pulmão e uma seção de biópsia de outros órgãos foram selecionadas. c, Pulmões, rins e baços dos pacientes 2-4 foram corados para proteína N viral. Para o paciente 2, o tecido pulmonar parecia negativo, enquanto edema (hash) e trombo em um pequeno vaso (asterisco) foram vistos. No rim, algumas células tubulares foram positivas para a proteína N, enquanto no baço apenas muito poucas células apresentaram coloração fraca. Paciente 3: o tecido pulmonar foi negativo para SARS-CoV-2 N, mostrando algum edema inicial e trombo em uma pequena artéria pulmonar (asterisco). No rim, as células do túbulo proximal foram positivas (seta), assim como algumas células da cápsula de Bowman do glomérulo (ponta de seta). Nenhuma proteína N foi observada no baço. Paciente 4: o tecido pulmonar parecia negativo, exceto por uma célula positiva no espaço intersticial. Um trombo é marcado por um asterisco; o hash marca o espaço alveolar. No rim, algumas células tubulares eram positivas para a proteína N, enquanto o glomérulo parecia negativo. No baço, um único agrupamento de células positivas foi detectado. A inserção mostra uma grande ampliação para ilustrar essa observação. Uma imagem representativa de uma seção de biópsia foi selecionada. Barra de escala, 100 µm.
No exame post-mortem, nós sistematicamente coramos diferentes órgãos para a proteína viral SARS-CoV-2 N usando dois anticorpos anti-N independentes, validados para imunohistoquímica em um modelo organoide de intestino SARS-CoV-2 70 (dados estendidos Fig. 8a ). No paciente 1, observamos coloração maciça de proteína N em pneumócitos que revestem o espaço alveolar, bem como macrófagos alveolares semelhantes a pneumonia aguda com dano alveolar difuso (Fig. 5b e Dados Estendidos Fig. 8b ), explicando a morte rápida deste paciente devido para insuficiência respiratória aguda 66. Além disso, algumas células epiteliais dos túbulos renais e alguns hepatócitos e colangiócitos coraram com proteína N positiva, enquanto o músculo cardíaco, nódulos linfáticos e baço não mostraram sinais de infecção por SARS-CoV-2 (Fig. 5b e Fig. Dados Estendidos 8b ). Nenhum sinal de proteína N viral claro foi detectado nos tecidos pulmonares dos outros três pacientes (Fig. 5c e Dados Estendidos Fig. 8b ) com um curso de doença mais longo, mas observamos macrotrombos (o asterisco na Fig. 5c ), que está em linha com as observações anteriores durante as fases posteriores do COVID-19 (refs. 4 , 71 ). Semelhante ao paciente 1 (Fig. 5b e Dados Estendidos Fig. 8b), encontramos a proteína N viral em todos os rins analisados, apoiando achados anteriores de um tropismo renal de SARS-CoV-2 (refs. 3 , 4 , 6 , 71 , 72 , 73 , 74 , 75 , 76 , 77 ) (Fig. . 5c e estendido de dados A Fig. 8b ). O rim humano obtido a partir de um exame post-mortem não COVID-19 serviu como um controle negativo para garantir uma coloração válida (dados estendidos Fig. 8c ). Nos baços, apenas os pacientes 2 e 4 mostraram algumas células infectadas espalhadas sem anormalidades grosseiras (Fig. 5c e Dados estendidos Fig. 8b)
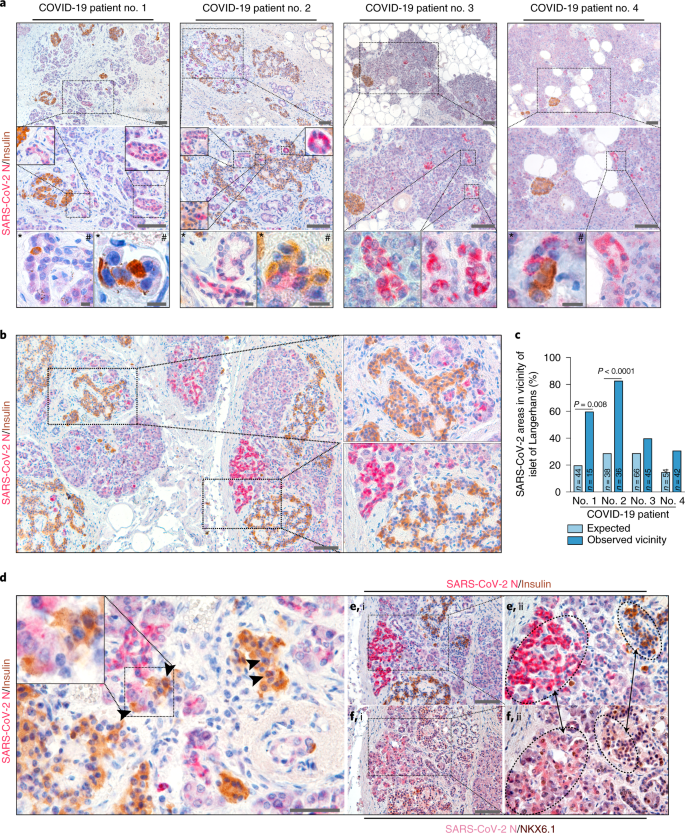
Em seguida, analisamos o envolvimento pancreático dos quatro pacientes. Durante o tratamento com COVID-19, observamos hiperglicemia nos pacientes 1, 2 e 3, acompanhada por uma demanda progressiva de insulina nos pacientes 2 e 3. A histopatologia pancreática revelou a presença da proteína N SARS-CoV-2, com número variável de células positivas em todos quatro pacientes, indicando uma infecção persistente durante COVID-19 grave independente do estágio inicial (paciente 1) ou tardio (paciente 2-4) da doença COVID-19 (Fig. 6a e Dados estendidos Fig. 8b ). Especificamente, a proteína N foi detectada em alguns dutos pequenos (CK19) e em células acinares simples ou agrupadas, mas foi negativa em uma amostra de controle não infectada (Fig. 6a e Dados estendidos Fig. 9), de acordo com o padrão de expressão da proteína de entrada viral (Fig. 1 e Extended Data Figs. 3 e 4 ). É importante notar que um paciente apresentou níveis elevados de lipase, indicando um certo grau de dano exócrino devido à infecção por SARS-CoV-2 (Tabela 1 ). As células positivas para a proteína N não foram espalhadas aleatoriamente pelo pâncreas humano, mas em vez disso, ocorreram em grupos de células infectadas, sugerindo disseminação viral localizada (Fig. 6a, b ). Para investigar a infecção de células β humanas, realizamos dupla coloração imunohistoquímica para a proteína N viral e insulina, mas observamos apenas algumas células duplamente positivas (Fig. 6a, close-ups marcados com um hash). No entanto, agrupamentos de células positivas para proteína N localizaram-se próximo às ilhotas de Langerhans, indicando certo grau de associação entre a infecção por SARS-CoV-2 e o compartimento endócrino. Isso foi quantificado por uma pontuação de vizinhança com base na distância entre os agrupamentos de células positivas para proteína N e insulina, e classificado em células com uma distância <100 µm ou ≥100 µm contra uma distância de referência calculada aleatoriamente. Em média, 51% dos agrupamentos de células infectadas com SARS-CoV-2 estavam localizados perto de ilhotas humanas, com um máximo significativo de 60% e 83% nos pacientes 1 e 2, bem como tendências semelhantes com 40% e 31% em pacientes 3 e 4, respectivamente (Fig. 6b, c ). Mais uma vez, algumas células positivas para insulina revelaram um sinal vermelho fraco de proteína N apontando para a disseminação da infecção (Fig.6d , pontas de seta). A morfologia de algumas das células claramente infectadas não se assemelhava à morfologia ductal, acinar ou endócrina, indicando um certo grau de plasticidade ocorrendo após a infecção. Isso está de acordo com a perda observada de hormônios e grânulos endócrinos, conforme sugerido por imunocoloração e imagens TEM de explantes de ilhotas infectados (Figs. 2 e 3 ). De fato, altos sinais de proteína N pareceram ir junto com baixa intensidade de coloração com insulina (Fig. 6d , close-up). Para abordar ainda mais isso, nós co-coramos para a proteína N e NKX6.1, que é exclusivamente expressa por células β no pâncreas adulto 55. Na verdade, detectamos células duplamente positivas N- / NKX6.1 em quatro de quatro pacientes, em estreita proximidade com as ilhotas de Langerhans e agrupamentos de células infectadas com SARS-CoV-2 (Fig. 6e, f e Extended Data Fig . 10 ). Partindo do pressuposto de que os clusters de células NKX6.1-positivas representam ou são derivadas do compartimento endócrino, estimamos a porcentagem de células endócrinas infectadas em média em 46% (intervalo 23-65%) (dados estendidos Fig. 10 ). Isso indica que a infecção de células β pode resultar em perda de hormônio, uma observação combinada por nossas análises ex vivo (Fig. 2 e Dados Estendidos da Fig. 5) Para concluir, a infecção pancreática SARS-CoV-2 pode ocorrer em casos graves de COVID-19, incluindo o compartimento exócrino e endócrino. No entanto, o tamanho da amostra ainda baixo, a qualidade do tecido subótima devido à necrose autolítica e os dados clínicos limitados impediram uma correlação com a função endócrina individual e o resultado nos pacientes.
a , Seções de tecido pancreático de quatro pacientes falecidos COVID-19 diferentes, coradas para proteína SARS-CoV-2 N (vermelho) e insulina (marrom). Os retângulos marcam as áreas de maior ampliação na próxima linha. As inserções mostram uma ampliação ainda maior para ilustrar padrões específicos. Os asteriscos marcam a ampliação das áreas manchadas fora das regiões ilustradas, mas correspondendo aos pacientes da respectiva coluna. A infecção ocorreu como agrupamentos N-positivos em todos os quatro pacientes com positividade de algumas células ductais e algumas células acinares. Células duplamente positivas de insulina e proteína N foram observadas em três de quatro pacientes (marcadas com um hash). Uma imagem representativa de uma seção de biópsia de cada paciente é exibida. b, As células N-positivas estão frequentemente localizadas nas proximidades das ilhotas de Langerhans ou mesmo misturadas em estruturas semelhantes a ilhotas com células positivas para insulina. A morfologia das células N-positivas frequentemente assemelha-se à morfologia não acinar / não ductal. Uma imagem representativa é derivada de uma seção de biópsia do paciente 1. c , Razão de vizinhança de regiões N-positivas (> 5 células positivas) localizadas perto de células endócrinas positivas para insulina (<100 µm de distância da célula endócrina) dividido por todas as N- regiões positivas (> 5 células positivas) revelam que o SARS-CoV-2 não está distribuído aleatoriamente pelo pâncreas, mas sim localizado próximo às estruturas endócrinas. Em média, 51% das regiões N-positivas estão localizadas perto de células endócrinas ou ilhotas. Para testes estatísticos,P <0,05, ** P <0,01, *** P <0,001, **** P <0,0001). d , Células virais N- e duplamente positivas para insulina são raras (pontas de seta), mas entrelaçadas nas ilhotas de Langerhans. O sinal de proteína N mais alto em um agrupamento de células ou mesmo em células individuais se correlaciona com o sinal de insulina mais baixo (close-up). e , f , Seções consecutivas foram coradas para insulina ( e ) ou NKX6.1 ( f), juntamente com a proteína N. Os retângulos em (i) marcam as áreas de maior ampliação (ii). As áreas tracejadas conectadas com setas de duas pontas marcam as regiões correspondentes que são altamente N positivas, insulina negativa e NKX6.1 positivo (esquerda) ou N negativo, insulina positiva e NKX6.1 positivo (direita). As imagens representativas são derivadas de uma seção de biópsia do paciente 2. Barras de escala, 100 µm; inserções, 10 µm.
Discussão
O curso da doença em pacientes com COVID-19 pode ser perturbado pelo diabetes mellitus de duas maneiras. Por um lado, o diabetes é um fator de risco para doença grave 3 , 16 , 17 , 18 , 19 , 20 , 21 , 22 e, por outro lado, a infecção por SARS-CoV-2 tem sido associada a alterações no metabolismo da glicose 3 , 16 , 24 , 29 . Especificamente, cetose e cetoacidose foram observadas durante e após a infecção por SARS-CoV-2, sendo ambas pelo menos indicadores clínicos indiretos de falta de insulina devido à perda ou mau funcionamento das células β 18 , 25, 26 , 27 , 28 , 29 , 30 . No presente estudo, nós: (1) dissecamos os padrões de expressão pancreática das proteínas de entrada viral do SARS-CoV-2 e (2) demonstramos permissividade das células β à infecção e replicação do SARS-CoV-2, que afeta (3) o subcelular morfologia e responsividade à glicose. Além disso, (4) demonstramos a presença do antígeno viral SARS-CoV-2 no pâncreas e, mais importante, nas células β positivas para NKX6.1 de pacientes falecidos COVID-19, alguns dos quais tinham diabetes pré-diagnosticado.
Analisamos ilhotas pancreáticas humanas para a expressão do fator de entrada de SARS-CoV-2 e observamos consistentemente a positividade ACE2 de células endoteliais intra-ilhotas, conforme relatado anteriormente 12 , 13 , 34 , 37 . Também demonstramos que as células endócrinas de ilhotas pancreáticas humanas expressam TMPRSS2, bem como as isoformas longas e curtas de ACE2, em linha com achados recentes 12 , 36 . No entanto, dados parcialmente conflitantes foram relatados sobre a expressão da isoforma ACE2 longa em células β humanas 13 , 34. Esses diferentes resultados experimentais podem ser atribuídos a variações intra e interindividuais no tecido do doador investigado, ou à utilização de anticorpos que possuem várias afinidades com as respectivas isoformas 12 , 13 , 34 , 36 . No presente estudo, usamos anticorpos estabelecidos em nossa análise imunohistoquímica que detecta as isoformas curta e longa e, além disso, realizamos uma extensa caracterização interna desses anticorpos e a metodologia aplicada 12 , 36 , 49 . Fignani et al. 12fornecem uma análise abrangente sobre a expressão de ACE2 em células β, incluindo detecção baseada em espectrometria de massa. Resumidamente, este estudo mostrou que a isoforma ACE2 curta é expressa em ilhotas pancreáticas humanas, onde é expressa preferencialmente em subconjuntos de células β produtoras de insulina 12 , 49 . É interessante também que Kusmartseva et al. relataram níveis detectáveis de mRNA de ACE2 em uma proporção de células endócrinas 13 . É importante ressaltar que outros fatores de entrada viral podem potencializar baixos níveis de expressão de ACE2 em células β. Especificamente, a neuropilina-1, um fator expresso nas células β pancreáticas 80 , e o receptor sequestrante B de lipoproteína de alta densidade tipo 1, também expresso em células endócrinas humanas81 , 82 , mostraram facilitar a infecciosidade do SARS-CoV-2 83 , 84 . No entanto, de acordo com o estado atual de conhecimento, a expressão de ACE2 continua sendo o principal determinante da entrada da SARS-CoV-2 e, portanto, do tropismo do organismo. Várias proteases podem preparar a proteína S do coronavírus, cuja expressão de TMPRSS2 pancreática também correspondeu ao nosso padrão de infecção de pacientes falecidos com COVID-19 6 , 8 , 37 , 85 , 86 . Descobrimos que ACE2 e TMPRSS2 colocalizam menos com marcadores de células δ e α. No entanto, esta frequência mais baixa não impede a infecção, conforme sugerido por dados anteriores 36. Reconhecemos que mais estudos são necessários para revelar os mecanismos exatos de entrada do SARS-CoV-2 nas células β e para avaliar os padrões de infecção de outras células endócrinas com mais detalhes. No entanto, independentemente de um potencial debate sobre a expressão da proteína de entrada viral, observamos infecção produtiva por SARS-CoV-2 de ilhotas cultivadas ex vivo usando o estado da arte, ensaios baseados em virologia molecular, fornecendo evidências para a presença de entrada funcional fatores.
Células β derivadas de células-tronco humanas pluripotentes podem ser infectadas por SARS-CoV-2 (ref. 87 ). Em células endócrinas de ilhotas de Langerhans cultivadas ex vivo humanas, detectamos proteínas virais, RNA viral e títulos virais infecciosos crescentes, e também visualizamos partículas de SARS-CoV-2 dentro de vacúolos na região perinuclear 56 por TEM. A observação mais impressionante foi um compartimento intermediário ER-Golgi ampliado e vacuolizado, semelhante às observações em células epiteliais intestinais, renais e das vias aéreas infectadas com SARS-CoV-2 56 , 57 , 58. As marcas da diferenciação endócrina, a saber, grânulos secretores, são deslocadas e significativamente reduzidas. No entanto, uma análise mais abrangente baseada em TEM em um ciclo completo de replicação viral em ilhotas humanas, bem como mais amostras de pacientes infectados, é necessária para tirar conclusões definitivas. No entanto, as observações do TEM estão de acordo com a secreção de insulina afetada observada em nosso estudo, embora tenhamos enfrentado variações experimentais nas quatro preparações de ilhotas investigadas. Digno de nota, as células β infectadas por enterovírus apresentam diminuição do GSIS e perda da estrutura corporal de Golgi 88 . Além disso, pode ocorrer desdiferenciação de células β simulando reversão para um estado progenitor acompanhado por diminuição da transcrição do gene específico de células β após o vírus 89mas também lesão química 90 . Nosso RNA-seq, análise de microscopia confocal e dados TEM estariam em linha com ambas as hipóteses, ou seja, estresse ER seguido de desgranulação e desdiferenciação de células β. No entanto, a lesão induzida pelo vírus pancreático também pode ser um driver de dano autopotenciador devido à inflamação, recrutamento de células imunes observadoras e, potencialmente, desenvolvimento de autoimunidade, que é específica para células β 67 . Na verdade, a infecção por SARS-CoV-2 provocou uma ampla assinatura de citocinas e ISGs atribuídos às respostas de IFN tipo I e II em ilhotas humanas. Recentemente, mostramos que as proteínas transmembrana induzida por IFN (IFITM) promovem a infecção por SARS-CoV-2 de células pulmonares humanas 91. Digno de nota, IFITM1–3 atingiu o topo entre os transcritos com regulação positiva em ilhotas humanas infectadas com SARS-CoV-2. Termos GO semelhantes foram relatados em organoides derivados do intestino após a infecção por SARS-CoV-2 58 , identificando tais respostas imunes desencadeadas intrinsecamente como uma característica geral em órgãos distintos durante COVID-19. Esses estudos globais de transcriptomas de ilhotas são, no entanto, limitados pelo design em massa, impedindo o acesso a transcriptomas específicos de tipo de célula e resolvidos por uma única célula 92. Finalmente, nossos resultados mostram que a replicação viral em ilhotas infectadas ex vivo foi eficientemente inibida por remdesivir usado como controle para prevenir a replicação do SARS-CoV-2. Esta inibição da replicação viral não foi associada a um resgate total na função das células β nem à restauração completa dos transcriptomas. Isso é provavelmente devido a um atraso na recuperação total das células β, que não pode ser alcançado no presente cenário experimental devido à deterioração das ilhotas em cultura ex vivo prolongada. No entanto, os efeitos do remdesivir sugerem que as alterações observadas são específicas da infecção por SARS-CoV-2.
A investigação do pâncreas de pacientes falecidos com COVID-19 revelou uma distribuição dispersa de grupos de células infectadas pelo pâncreas em todos os quatro pacientes, mais visíveis no compartimento exócrino, mas com proximidade às ilhotas de Langerhans. Tal padrão pode indicar disseminação para células pancreáticas vizinhas originadas de algumas células infectadas, potencialmente atingidas por partículas virais diretamente através da corrente sanguínea durante a viremia temporária, ocorrendo tipicamente em COVID-19 grave (refs. 93 , 94 ). Kusmartseva et al. também investigou a expressão da proteína N em pacientes falecidos COVID-19, mas não conseguiu detectar a co-expressão com insulina 13. Em nosso estudo, a detecção de células N- / insulina-duplamente positivas foi tecnicamente desafiadora e rara em frequência. No entanto, com base em nossas observações em ilhotas cultivadas ex vivo, a co-coloração da proteína N viral com o marcador de linhagem de células β NKX6.1 confirmou a infecção por SARS-CoV-2 em agrupamentos de células que expressam NKX6.1 em todos os pacientes investigados com COVID- 19 Notavelmente, a expressão pancreática de NKX6.1 é única porque nenhum outro fator de transcrição é restrito exclusivamente a células β dentro do pâncreas adulto 55. Como também observamos essas células hormonais negativas nas preparações de ilhotas humanas, isso sugere que a infecção por SARS-CoV-2 pode perturbar a positividade do hormônio por citocina e / ou estresse ER, seguido por desgranulação e desdiferenciação de células β. Uma análise mais aprofundada é necessária para compreender totalmente o mecanismo subjacente e é importante observar que a perda de hormônio na infecção complica essas avaliações experimentais. Especificamente, a insulina pode não ser um marcador adequado para mostrar co-localização com proteínas virais. Assim, estimar as taxas de infecção das respectivas células, em ilhotas ou tecidos pancreáticos, permanece um desafio. O padrão preciso de infecção de SARS-CoV-2 em subpopulações de ilhotas humanas ainda é garantido, e se o número de células infectadas com SARS-CoV-2 e a perda de identidade associada de células β são suficientes para afetar a função endócrina em ilhotas saudáveis e doentes ainda precisam ser determinados. Consequentemente, nossos resultados levantam a questão de saber se o SARS-CoV-2 perturba diretamente a integridade das células β e potencialmente leva à desregulação endócrina e causa DMT1 autoanticorpo negativo, conforme relatado em estudos clínicos recentes após infecção por SARS-CoV-29 , 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 27 , 28 , 29 , 30 . Alternativamente, a infecção por SARS-CoV-2 pode ser um fator precipitante de diabetes mellitus autoimune mediado, surgindo mesmo anos após a recuperação 1 , 22 , justificando a necessidade de acompanhamento de longo prazo de pacientes com COVID-19 33 . Embora, por exemplo, Coxsackie B4 ou infecções congênitas pelo vírus da rubéola possam desencadear o desenvolvimento de DM167 , 95 , 96 , o insulto de células β mediado por vírus se apresenta de forma heterogênea em diferentes vírus, por exemplo, a infecção de células β por enterovírus pode levar a: (1) morte celular acompanhada por aumento da proliferação em células β vizinhas não infectadas , (2) produção e secreção de insulina prejudicadas, ou (3) desdiferenciação de células β 89 , 97 . Coletivamente, se a infecção por SARS-CoV-2 desencadeia uma resposta imune prejudicial ou reduz diretamente a função das células β, afetando assim o sistema endócrino, precisa ser avaliado em estudos futuros.
A infecção dos ductos pancreáticos e células acinares levanta ainda a questão de saber se há uma correlação com os aumentos dos níveis de lipase observados e pancreatite edematosa aguda em pacientes com COVID-19 com pneumonia associada à SARS-CoV-2. Pancreatite aguda também foi relatada como ocorrendo em COVID-19 (refs. 5 , 22 , 23 , 98 ). Estudos recentes relataram pancreatite aguda em 12,6% de toda a coorte e 32,5% em pacientes críticos 23 . No entanto, não está claro se a pancreatite aguda, uma doença potencialmente fatal porque causa deterioração em pacientes graves, ocorre como um efeito colateral ou uma consequência direta da infecção pancreática por SARS-CoV-2.
A análise de outros órgãos de pacientes falecidos com COVID-19 revelou coloração robusta dos rins, enquanto o baço não estava infectado de forma consistente. O tecido pulmonar apresentou diferentes padrões de infecção, que podem corresponder a diferenças na apresentação temporal e espacial da doença 71 , 99 . Não podemos excluir a infecção em outras regiões do pulmão ou baço devido ao viés de amostragem durante o exame post-mortem e ao baixo tamanho da amostra. Em particular, a questão do tropismo renal permanece em discussão. Digno de nota, há relatos de que os rins são altamente suscetíveis à infecção por SARS-CoV-2 3 , 6 , 71 , 73 , 74 , 75 , 76 ,77 , 78 , 79 , enquanto outros não conseguiram detectar elementos virais nos rins 78 , 100 , 101 , 102 , 103 . Essas discrepâncias revelam que nosso conhecimento sobre (1) as frequências e (2) a associação com doenças pré-existentes e (3) o impacto clínico das infecções extrapulmonares por SARS-CoV-2 permanece incompleto. Existem várias explicações para esses achados conflitantes, como a heterogeneidade espacial e temporal da disseminação viral, restrições técnicas como a preservação da amostra e os ensaios de detecção usados. Biópsias de tecido guiadas por ultrassom e resolvidas com o tempo podem contornar as limitações da análise de endpoint em estudos post mortem.
Coletivamente, demonstramos que os compartimentos exócrino e endócrino do pâncreas são suscetíveis à infecção produtiva de SARS-CoV-2, que pode perturbar a integridade das células β. O mecanismo de dano induzido por vírus e se a infecção tem uma consequência direta para a homeostase da glicose ou pode até mesmo desencadear diabetes mellitus continuam em discussão e merecem estudos futuros.
Métodos
Drogas
Remdesivir foi obtido de Selleck Chemicals (catálogo no. S8932).
Cultura de células
Células Vero E6 ( Cercopithecus aethiops – rim epitelial derivado, American Type Culture Collection (ATCC)) foram cultivadas em meio Eagle modificado por Dulbecco (DMEM, Gibco) que foi suplementado com 2,5% de soro fetal de bezerro inativado pelo calor (FCS), 100 U ml −1 de penicilina, 100 μg.ml −1 de estreptomicina, 2 mM de L- glutamina e 1 mM de piruvato de sódio. As células Calu-3 (adenocarcinoma epitelial de pulmão humano, gentilmente cedido pelo Professor Frick, Ulm University) foram cultivadas em meio essencial mínimo Eagle (MEM, Sigma-Aldrich, nº de catálogo M4655) suplementado com 10% de FCS, 100 U ml −1 de penicilina, 100 μg ml −1de estreptomicina, piruvato de sódio 1 mM e 1 × aminoácidos não essenciais. As células EndoC-βH1 (Univercell Biosolutions) foram cultivadas em poços de cultura revestidos (2 µg ml -1 de fibronectina, matriz extracelular a 1%) em meio OPTIβ1 (Univercell Biosolutions) de acordo com o protocolo do fabricante. As células endoteliais da veia umbilical humana foram cultivadas em meio de crescimento de células endoteliais (Merck). As células foram cultivadas a 37 ° C em 5% de CO 2incubadora umidificada. Para a diferenciação de organóides intestinais derivados de células-tronco, a linha de células-tronco embrionárias humanas (hESC) HUES8 (Universidade de Harvard) foi usada com permissão do Instituto Robert Koch de acordo com ’79. Genehmigung nach dem Stammzellgesetz, AZ 3.04.02 / 0084 ‘. Para diferenciação, 300.000 células por poço foram semeadas em placas de 24 poços revestidas com Matrigel com fator de crescimento reduzido (Corning) em mTeSR1 com inibidor ROCK 10 µM. No dia seguinte, a diferenciação foi iniciada por lavagem com solução salina tamponada com fosfato (PBS) e adição de meio de diferenciação d0 (meio BE1 (MCDB131 (Invitrogen), L- glutamina 2 mM (Gibco), 1,174 g l- 1 de bicarbonato de sódio (Sigma-Aldrich ), 0,8 gl −1de glicose (Sigma-Aldrich), albumina de soro bovino livre de ácidos graxos 0,1% (BSA, Proliant)) com 100 ng ml −1 de Activina A (R & D / PeproTech) e 2 µM de CHIR99021 (Axon MedChem). Após 24 he 48 h, o meio foi trocado para meio d1 / d2 (BE1 com 100 ng ml −1 de ativina A). A partir do dia 3, o meio foi trocado diariamente para meio de formação de intestino posterior médio fresco (RPMI-1640 (Gibco), 2% de FCS (Biochrom), 2 mM de L- glutamina (Gibco), 1% de penicilina-estreptomicina (Sigma-Aldrich ), 10 ng ml −1 de BMP4 (Peprotech), 100 ng ml −1de fator de crescimento de fibroblasto básico (Novoproteína), CHIR99021 3 µM (Cayman Chemical Co.)). A partir do dia 7, as estruturas começaram a flutuar no meio e foram coletadas e plaqueadas em placas Nunclon de 48 poços (Sigma-Aldrich) em cúpulas Matrigel (20–40 µl de Matrigel por cúpula). Em seguida, 350 µl de meio de crescimento intestinal (DMEM F12 (Gibco), suplemento 1 × B27 (Thermo Fisher Scientific), 2 mM de L- glutamina, 1% de penicilina-estreptomicina (Sigma-Aldrich), 40 mM de 4- (2-hidroxietil ) Solução de ácido -1-piperazina-etanossulfônico (Hepes) (Sigma-Aldrich), 3 µM de CHIR99021, 200 nM de LDN-193189 (Sigma-Aldrich) e 100 ng ml -1 de fator de crescimento epitelial humano (Novoproteína)) foi adicionado por bem. O meio foi mudado duas vezes por semana.
Cepas de vírus e propagação de vírus
O isolado viral BetaCoV / Netherlands / 01 / NL / 2020 (catálogo no. 010V-03903) da variante pandêmica D614G foi obtido do European Virus Archive global e propagado em células Vero E6. Para tanto, as células 70% confluentes em frascos de cultura de células de 75 cm² foram inoculadas com 100 µl de SARS-CoV-2 isolado em 3,5 ml de meio sem soro contendo 1 µg ml −1 de tripsina. As células foram incubadas durante 2 h a 37 ° C, antes da adição de 20 ml de meio contendo Hepes 15 mM. As células foram incubadas a 37 ° C e o sobrenadante colhido quando um forte efeito citopático era visível. Os sobrenadantes foram centrifugados por 5 min a 1.000 gpara remover resíduos celulares e, em seguida, divididos em alíquotas e armazenados a −80 ° C como estoques de vírus. O título do vírus infeccioso foi determinado como unidades formadoras de placas em células Vero E6, que foi usado para calcular a multiplicidade de infecção (MOI).
Isolamento de RNA e RT-qPCR
O RNA viral das células foi isolado usando o QIAGEN RNeasy Plus Mini Kit (catálogo no. 74136) e o RNA de sobrenadantes usando o QIAGEN Viral RNA Mini Kit (catálogo no. 52906) conforme descrito pelo fabricante. As células foram lisadas em 600 μl de tampão RLT Plus contendo 1% de β-mercaptoetanol, submetidas a vórtice por 30 s e então congeladas a −20 ° C até posterior isolamento. Para os sobrenadantes, 140 μl foram misturados com 560 μl de tampão AVL, agitado em vórtice e congelado como acima. A transcrição reversa (RT) -qPCR de células ou sobrenadantes foi realizada com conjuntos de iniciadores direcionados a SARS-CoV-2 N (nucleocapsídeo) ou ORF1b-nsp14 (refs. 108 , 109) usando TaqMan Fast Virus 1-Step Master Mix (Thermo Fisher Scientific, catálogo nº 4444436) e um sistema StepOnePlus Real-Time PCR (formato de 96 poços, modo rápido). A expressão de ACE2 e TMPRSS2 foi avaliada usando ensaios TaqMan (Thermo Fisher Scientific, catálogo nos. Hs01085333_m1 e Hs01122322_m1, respectivamente). SARS-CoV-2-RNA sintético (Twist Bioscience, catálogo no. 102024 ou ATCC, catálogo no. VR-3276SD) foi usado como um padrão para obter números de cópias virais para quantificação de RNA viral em sobrenadantes. Para RNA isolado de células, gliceraldeído 3-fosfato desidrogenase (GAPDH) foi usado como um controle endógeno (Applied Biosystems, nº de catálogo 4310884E) para calcular a expressão relativa. O limite para detecção foi definido no ciclo limite, C t, de 35. Todas as reações foram realizadas em duplicado.
Conjuntos de primer:
Alvo N:
Iniciador direto (HKU-NF): 5′-TAATCAGACAAGGAACTGATTA-3 ′
Primer reverso (HKU-NR): 5′-CGAAGGTGTGACTTCCATG-3 ′
Sonda (HKU-NP): 5′-FAM-GCAAATTGTGCAATTTGCGG-TAMRA-3 ′
ORF1b-nsp14 alvo:
Iniciador direto (HKU-ORF1b-nsp14F): 5′-TGGGGYTTTACRGGTAACCT-3 ′
Iniciador reverso (HKU- ORF1b-nsp14R): 5′-AACRCGCTTAACAAAGCACTC-3 ′
Sonda (HKU-ORF1b-nsp141P): 5′-FAM-TAGTTGTGATGCWATCATGACTAG-TAMRA-3 ′.
Titulação de endpoint TCID 50
Para determinar a dose infecciosa 50 da cultura de tecidos (TCID 50 ), as amostras do sobrenadante foram diluídas em série e usadas para inocular células Vero E6. Para este fim, 20.000 células Vero E6 foram semeadas por poço em placas de fundo plano de 96 poços em 100 µl de meio e incubadas durante a noite antes da adição de 62 µl de meio fresco. Em seguida, 18 µl de amostra titulada por cinco ou dez vezes foram usados para inoculação em triplicado. As células foram incubadas por 4-6 dias e monitoradas quanto ao efeito citopático. O TCID 50 por ml foi calculado de acordo com Reed e Muench 110 .
Cultura de ilhotas
Quatro preparações de ilhotas pancreáticas humanas foram obtidas no Alberta Diabetes Institute IsletCore da Universidade de Alberta, chefiado por PE MacDonald. O material das ilhotas foi derivado de órgãos de doadores excedentes usados para transplante clínico e com o consentimento informado por escrito para pesquisa (aprovação nº Pro00013094). O primeiro doador (pureza de 95%), feminino, 31 anos, apresentava índice de massa corporal (IMC) de 20,3 e sem histórico de diabetes (hemoglobina glicada (HbA1c): 4,8%). O segundo doador (50% de pureza), do sexo feminino, com 55 anos, tinha um IMC de 29,1 e nenhum histórico de diabetes (HbA1c: não fornecido). O terceiro doador (40% de pureza), homem, 58 anos, tinha IMC de 31 e sem histórico de diabetes (HbA1c: 5,6%). O quarto doador (90% de pureza), feminino, com 53 anos de idade, tinha IMC de 33,2 e sem histórico de diabetes (HbA1c: 6,1%). Este estudo também foi aprovado pelo ‘Ethikkommission TUM’ no. 394 / 20S. Após o transporte, as ilhotas foram lavadas em meio de cultura e semeadas em placas de fixação ultrabaixa de 12 poços (Corning) a uma densidade de 400 ilhotas por poço. Os experimentos de infecção foram iniciados 48 horas após a recuperação. Ilhotas pancreáticas foram cultivadas em CMRL1066 (Gibco) suplementado com 10% de soro humano (Sigma-Aldrich), 2 mML- glutamina (Sigma-Aldrich), 1% penicilina-estreptomicina (Sigma-Aldrich) e tampão Hepes 25 mM (Sigma-Aldrich). O meio foi substituído a cada dois dias.
Infecção de ilhotas
Ilhotas pancreáticas do doador 1 em 800 µl de meio foram infectadas pela adição de 200 µl de inóculo de vírus, resultando em um MOI de ~ 1. Para atingir taxas de infecção mais altas, as ilhotas de doadores 2-4 foram pré-incubadas com TrypLE por 5 min, a reação interrompida pela adição de DMEM / F12 contendo BSA, o sobrenadante descartado e as ilhotas infectadas com um MOI de ~ 2,5. Após 3 horas de incubação, as ilhotas foram lavadas três vezes e cultivadas em 1 ml de meio por 24 poços ou 4 ml por 6 poços, que foi em um ambiente suplementado com 5 µM de remdesivir. Nos pontos de tempo indicados, 0,5 ou 2 ml de sobrenadante do meio foram coletados para análise de RT-qPCR e TCID 50 e o meio foi recarregado respectivamente.
GSIS estático
GSIS estático foi realizado 3 dias após a infecção. Ilhotas pancreáticas foram lavadas em tampão de bicarbonato Krebs-Ringer (KRBH) contendo BSA 0,1% e incubadas por 1 h em KRBH com glicose 2 mM. Após 1 h, as ilhotas foram lavadas em KRBH / BSA e ressuspensas em um tampão contendo glicose 2 mM. Após 1 h de incubação, o sobrenadante foi retirado e armazenado para quantificação da insulina; as ilhotas foram lavadas em KRBH e incubadas em KRBH com glicose 20 mM por mais uma hora. O sobrenadante foi retirado e armazenado para quantificação da insulina. O procedimento GSIS foi realizado para uma amostra de ilhotas (doador 1) e amostras de três ilhotas (doadores 2 e 4). Os sobrenadantes do GSIS estático foram analisados em duplicata com o kit ELISA de insulina ultrassensível (Alpco), de acordo com as instruções do fabricante. De importância, a estimulação de uma amostra de ilhota não infectada (doador 2) com glicose 2 mM não resultou em secreção de insulina detectável. Os valores foram normalizados para números de células ou conteúdo de proteína (quantificado por ensaio BCA, Thermo Fisher Scientific, catálogo no. A53227) e a indução de vezes da secreção de insulina (estimulação de glicose 20 mM em comparação com glicose 2 mM) foi calculada para cada amostra.
TEM de ilhotas
A preparação da amostra para TEM foi feita conforme descrito anteriormente 111 . Para tanto, as ilhotas pancreáticas foram lavadas uma vez com PBS e fixadas com glutaraldeído 2,5% contendo sacarose 1% em tampão fosfato, pH 7,3. As amostras foram lavadas com PBS e fixadas posteriormente em tetróxido de ósmio aquoso a 2%. Após desidratar as amostras em uma série graduada de 1-propanol, elas foram coradas em acetato de uranila a 2% e incluídas em Epon. Seções ultrafinas (80 nm) foram coletadas em grades de cobre TEM, contrastadas com citrato de chumbo 0,3% por 1 min e fotografadas em um Jeol TEM 1400 a 120 kV.
Histologia de seções de tecido pancreático saudáveis e cultura ex vivo de ilhotas humanas
Seções de pâncreas humano foram fornecidas pelo departamento de patologia da Universidade de Ulm. A integridade do tecido pancreático não neoplásico foi aprovada por um patologista certificado (TFEB). Os experimentos foram conduzidos de acordo com as diretrizes do Comitê de Ética do Conselho Geral de Medicina da Federação e aprovados pelo Comitê de Ética da Universidade de Ulm (votação para utilização de material humano arquivado 03/2014). As seções foram desparafinizadas, reidratadas e submetidas à recuperação de antígeno mediada por calor em tampão Tris, pH 9, ou tampão citrato, pH 6. O tecido foi permeabilizado com 0,5% Triton X-100 por 30 min em temperatura ambiente e corado durante a noite com anticorpos primários (Dados estendidos Fig. 1) em diluente de anticorpo (Zytomed) em uma câmara úmida a 4 ° C. Após a lavagem com PBS-Tween 0,05% (PBS-T), as lâminas foram incubadas com anticorpos secundários (Alexa Fluor IgG H + L, Invitrogen, 1: 500) e 500 ng ml −1 de 4 ′, 6-diamidino-2- fenilindol (DAPI) em diluente de anticorpo por 90 min em uma câmara úmida em temperatura ambiente. Após lavagem com PBS-T e água, as lâminas foram montadas com Fluoromount-G (Southern Biotech). Para experimentos de bloqueio de anticorpos, o anticorpo primário ACE2 com peptídeo de bloqueio (dados estendidos Fig. 1 ) em sete vezes a quantidade de proteína em excesso foi incubado por 30 min em temperatura ambiente em diluente de anticorpo antes da coloração.
Para exame histológico, as ilhotas pancreáticas foram fixadas em paraformaldeído a 4% (PFA) durante a noite. Em seguida, foram incubados em sacarose 1 M em PBS durante a noite e incluídos no composto de congelamento OCT (Tissue-Tek). Os crioblocos foram seccionados a 7 µm e as lâminas armazenadas a −80 ° C. A coloração por imunofluorescência foi realizada de forma semelhante aos cortes de parafina, exceto para as etapas de desparafinização, reidratação e recuperação do antígeno, que foram omitidas. A lavagem entre as etapas de coloração foi realizada com Triton X-100 0,1% em PBS.
Os controles negativos foram realizados com controles de imunoglobulina (Ig) G ou soro policlonal irrelevante (anti- Mycobacterium tuberculosis ) para anticorpos policlonais, respectivamente. A ausência de coloração de fundo confirmou a especificidade dos anticorpos primários. Imagens confocais de varredura a laser foram adquiridas usando o Zeiss LSM710. Alternativamente, algumas imagens mostradas nas Figs. 1 e 2foram adquiridos usando um Leica TCS SP8 equipado com uma objetiva de imersão HC PL APO CS2 63 × / 1,2 WATER. As imagens foram adquiridas no modo de varredura sequencial como seções aéreas confocais simples usando detectores HyD e a seguinte faixa de detecção: Ex: 405 nm, Em: 430–470 nm; Ex: 488 nm, Em: 500–535 nm; Ex: 561, Em: 571–620 nm. A colocalização foi quantificada usando a ferramenta de quantificação Coloc2 de Fiji e configurações padrão. A fração de células positivas para a proteína N SARS-CoV-2 (Fig. 2c ) foi quantificada em ilhotas infectadas de doadores 2 e 3 (dias 3 e 5), coradas para a proteína N SARS-CoV-2. As células positivas para a proteína N foram contadas em sete (dia 3) e seis (dia 5) aglomerados de ilhotas positivos para a proteína N e a fração da população total de células é mostrada na Fig. 2c . Ilhotas não infectadas de cada doador (n = 4) serviu como controle.
Imagens de ilhotas infectadas com SARS-CoV-2 do doador 3 (dia 5) coradas para NKX6.1 e proteína N de SARS-CoV-2 (dados estendidos Fig. 6d ) foram adquiridas usando um ApoTome Zeiss. Células positivas para NKX6.1 e células positivas para proteína N foram quantificadas em 11 grupos de ilhotas positivas para proteína N e a fração de células NKX6.1 e duplamente positivas para proteína N da população total de células positivas para NKX6.1 é mostrada em Dados estendidos Fig. 6e .
Imunohistologia de seções de tecido de pacientes infectados com SARS-CoV-2
As amostras de tecido foram fornecidas pelo banco de tecidos do Centro Alemão de Pesquisa de Infecções (DZIF, Heidelberg, Alemanha, aprovação S242 / 2020) e do Instituto de Patologia da Universidade de Ulm. As características dos pacientes são relatadas na Tabela 1. As colorações imunohistoquímicas duplas foram realizadas por incubação sequencial com anticorpos de diferentes espécies contra o nucleocapsídeo (proteína N) do SARS-CoV-2 (camundongo, 1:50 in situ, 1: 100 in vitro) e contra a insulina como marcador para β- endócrino células (coelho, 1: 10.000 in situ, 1: 5.000 in vitro), NKX6.1 (camundongo, 1:75 in situ) e CK19 (camundongo, 1: 100 in situ) como marcadores ductais. A recuperação do antígeno foi realizada por tratamento das lâminas em tampão citrato, pH 6,1, em vaporizador por 25 min. A ligação específica ao antígeno foi assegurada por incubação de todos os anticorpos usados por 30 min em temperatura ambiente. A detecção de anticorpos foi feita pelo sistema de detecção de fosfatase alcalina / RED (Dako REAL Detection System) para proteína N e sistema de cromogênio DAB (Dako EnVision HRP DAB System) para insulina e NKX6.1. Para imagens,
A distância dos agrupamentos de células positivas para a proteína N às células positivas para a insulina (pontuação de vizinhança) foi quantificada usando o software de imagem Leica LAS-X e classificada em células com uma distância <100 µm ou ≥100 µm. Para permitir o teste estatístico, uma distância de referência foi gerada (número esperado de células na proximidade da ilhota). Para isso, as células foram selecionadas aleatoriamente usando o software de imagem LAS-X e a respectiva distância dessas células de referência às ilhotas foi quantificada. Para o teste estatístico, o teste exato de Fisher foi aplicado aos números absolutos das tabelas de contingência. Para ilustração, apenas a fração de células, não os números absolutos de células, com uma proximidade <100 µm para ilhotas C-pep-positivas é exibida (* P <0,05, ** P <0,01, *** P <0,001, * ***P <0,0001).
Partindo do pressuposto de que os agrupamentos de células positivas para NKX6.1 (> 10 núcleos positivos nas proximidades, com morfologia de ilhotas preservada e perturbada) representam ou são derivados do compartimento endócrino, estimamos o número de agrupamentos infectados em áreas não necróticas dos espécimes (definido como morfologia acinar preservada). Para este fim, 20 agrupamentos atribuídos aleatoriamente foram avaliados quanto à expressão da proteína N e agrupamentos contendo mais de cinco células duplamente positivas foram considerados infectados por SARS-CoV-2. Uma análise mais detalhada e quantificação foram impedidas por grandes áreas de tecido necrótico em tecido pancreático post mortem (porcentagem de área necrótica fornecida na Fig. 10b de Dados Estendidos ).
RNA-seq
Preparação da biblioteca NGS
Para a preparação de bibliotecas de sequenciamento de próxima geração (NGS) a partir de amostras de baixa entrada de isolados de RNA celulares, seguimos o protocolo 112 Smart-seq2 . As concentrações da biblioteca foram quantificadas com o sistema Qubit 2.0 Fluorometric Quantitation (Life Technologies) e a distribuição de tamanho foi avaliada usando o Experion Automated Electrophoresis System (Bio-Rad). Para o sequenciamento, as amostras foram diluídas e agrupadas em bibliotecas NGS em quantidades equimolares.
Sequenciamento e processamento de dados brutos
Bibliotecas de perfil de expressão foram sequenciadas em um instrumento HiSeq 4000 (Illumina) em 50 bp, modo de extremidade única. Chamadas de base, fornecidas pelo software de análise em tempo real (RTA, v.2.7.7) (Illumina), foram subsequentemente convertidas em formato BAM multiplexado e não alinhado antes da desmultiplexação em arquivos BAM não alinhados e específicos de amostra. Para o processamento de dados brutos fora dos instrumentos , foram utilizados programas customizados, baseados nas ferramentas Picard (v.2.19.2) 113 .
Análise de transcriptoma
As leituras NGS foram mapeadas para a montagem GRCh38 do Genome Reference Consortium via ‘Alinhamento de transcritos emendados para uma referência’ (STAR, v.2.7.5a) 114 usando a anotação de transcrição Ensembl ‘básica’ de v.e100 (abril de 2020) como transcriptoma de referência. Como o tipo de montagem hg38 da Universidade da Califórnia, Santa Cruz (UCSC) Genome Browser foi preferido para processamento de dados downstream com pacotes Bioconductor por razões inteiramente técnicas, a anotação de transcrição Ensembl teve que ser ajustada para nomes de região de sequência do navegador de genoma UCSC. O STAR foi executado com opções sugeridas pelo projeto ENCODE. NGS alinhado lê sobrepondo recursos de transcrição Ensembl foram contados com a função Bioconductor (3.11) GenomicAlignments :: sumizeOverlaps () (1.24.0) 115, levando em consideração que o protocolo Smart-seq2 não é específico de strand. As contagens em nível de transcrição foram agregadas às contagens em nível de gene e o pacote Bioconductor DESeq2 (v.1.28.1) 104 foi usado para testar a expressão diferencial com base em um modelo usando a distribuição binomial negativa.
Uma análise exploratória inicial incluiu análise de componente principal, escala multidimensional, distância de amostra e gráficos de mapa de calor de expressão, todos anotados com variáveis usadas na modelagem de expressão (CRAN ggplot2 v.3.3.2 (ref. 116 ), Bioconductor ComplexHeatmap v.2.4.3 ( ref. 117 )), bem como gráficos de vulcão (Bioconductor EnhancedVolcano v.1.6.0 (ref. 118 )). Os mapas de calor de distância de amostra foram obtidos por meio do pacote CRAN pheatmap 119calculando a matriz de distância euclidiana (via R stats :: dist ()) dos valores de contagem normalizados DESeq2 após a transformação de estabilização de variância no modo ciente de modelo (via DESeq2 :: varianceStabilizingTransformation) e subsequente agrupamento hierárquico com o método de ligação completo (via R stats :: hclust ()). Resultados biologicamente significativos foram extraídos do modelo, os valores de log 2 (variação) (log 2 (FC)) foram reduzidos com o pacote CRAN ashr (v.2.2-47) 120 e os valores de P foram ajustados usando a ponderação de hipótese independente do biocondutor (IHW, v.1.16.0) pacote 121 . As listas de genes resultantes foram anotadas, filtradas para genes significativamente diferencialmente regulados para cima e para baixo ( P ajustado <0,1 e log 2 (FC)> | 1 |), e foram independentemente submetidos a análises de sobre-representação. Enquanto g: Profiler [v .: e100_eg47_p14_7733820] 105 foi aplicado para testar o enriquecimento de genes significativamente diferenciados contra conjuntos de genes em bancos de dados comuns, o EnrichR permitiu a expansão para termos de doenças relacionadas a COVID-19 (Enrichr) 106 , 122 . Para GSEA, a ferramenta de desktop gsea 107 foi usada (GSEA v.4.0.3) com a configuração de ‘Tipo de permutação’ para ‘gene_set’ e ‘Métrica para classificação de genes’ para ‘Signal2Noise’. O enriquecimento foi testado contra conjuntos de genes de marca registrada de MSigDB 7.2 (ref. 123 ) e um cluster de genes de células β adicional derivado de Ackermann et al. 66. As pontuações de enriquecimento foram verificadas com a ferramenta GSEA Preranked usando log 2 (FC) como parâmetro de classificação, produzindo resultados altamente semelhantes.
Lisados de células e tecidos inteiros
Para determinar a expressão de proteínas celulares, o tecido pancreático congelado foi pulverizado com um pilão e almofariz resfriado com nitrogênio líquido e diretamente transferido para um tubo em gelo seco para reduzir a degradação da proteína. Amostras de tecido pulverizado, bem como pellets celulares, foram lavados em PBS e lisados em tampão de lise Western blot (NaCl 150 mM, Hepes 50 mM, ácido etilenodiaminotetracético 5 mM, NP-40 0,1%, Na 3 VO 4 500 μMe 500 μM de NaF, pH 7,5) suplementado com inibidor de protease (Roche). Após 5 min (lisado de células) ou 20 min (lisado de tecido) de incubação em gelo, as amostras foram centrifugadas (4 ° C, 20 min, 14.000 rpm) para remover os resíduos celulares. O sobrenadante foi transferido para um tubo novo, a concentração de proteína foi medida usando o ensaio BCA (Thermo Fisher Scientific, catálogo no. A53227) e a concentração foi ajustada usando tampão de lise Western blot.
Eletroforese em gel de dodecilsulfato de sódio-poliacrilamida e imunoblotting
Para western blotting, lisados de células inteiras foram misturados com 4 × tampão de carregamento de amostra de proteína (LI-COR, a uma diluição final de 1 ×) suplementado com 10% de β-mercaptoetanol (Sigma-Aldrich), aquecido a 95 ° C por 10 min, separados em NuPAGE 4–12% Bis – Tris Gels (Invitrogen) por 90 min a 110 V e manchados em membranas Immobilon-FL poli (fluoreto de vinilideno) (Merck Millipore). A transferência foi realizada em uma tensão constante de 30 V por 30 min. Após a transferência, a membrana foi bloqueada com caseína a 1% em PBS (Thermo Fisher Scientific, catálogo nº 37528). As proteínas foram coradas usando anticorpos primários (dados estendidos Fig. 1) contra ACE2 (1: 1.000, Abcam), TMPRSS2 (1: 1.000, Abcam) e GAPDH (1: 5.000, Abcam, Bio-Rad) e anticorpos secundários marcados com corante infravermelho (LI-COR IRDye; Bio-Rad StarBright ) Western blots não cortados e não processados são fornecidos com os dados de origem .
Imunofluorescência de células EndoC-βH1
As células EndoC-βH1 foram semeadas em uma lâmina IBIDI de 8 µm. Após 48 h, as células foram fixadas com PFA a 1% por 15 min em temperatura ambiente. As células foram tratadas com 0,1 M de glicina por 10 min em temperatura ambiente, seguido por permeabilização com 0,25% de Triton X-100 em PBS por 10 min e subsequente bloqueio com 3% de BSA em 0,1% de Triton X-100 por 30 min em temperatura ambiente. Após o bloqueio, os anticorpos primários foram adicionados, diluídos em 5% de soro normal de burro em 0,1% de Triton X-100 em PBS e incubados por 2 h em temperatura ambiente. As células foram lavadas duas vezes e incubadas com anticorpos secundários e DAPI em 5% de soro de burro normal em 0,1% de Triton X-100 em PBS por 1 h em temperatura ambiente no escuro. Para experimentos de bloqueio de ACE2, anticorpo primário ACE2 com e sem peptídeo de bloqueio (Dados estendidos Fig. 1) a sete vezes a quantidade de proteína em excesso foi incubada por 30 min em temperatura ambiente no respectivo tampão.
Cultura de células e coloração de imunofluorescência de HAECs
HAECs diferenciadas foram geradas conforme descrito por Winkelmann et al. 124 . Todos os experimentos foram realizados com a aprovação do comitê de ética da Faculdade de Medicina de Hannover (projeto no. 2701-2015). Resumidamente, 3,5 × 10 4células epiteliais basais obtidas de vários doadores foram semeadas em filtros Transwell de 6,5 mm revestidos com colágeno (Corning Costar). Ao atingir a confluência (~ 48 h), o meio do lado apical foi removido (levantamento de ar) e o lado basolateral substituído por meio de diferenciação de interface ar-líquido (DMEM-H e LHC Basal (1: 1), Thermo Fisher Scientific, suplementado com Pacote de suplemento do meio de crescimento de células epiteliais das vias aéreas, Promocell). Para evitar o acúmulo de muco, as células foram lavadas com PBS a cada 3 dias a partir do dia 14. Em seguida, 25-30 d após o levantamento de ar, as células foram fixadas com PFA 4% em PBS por 30 min e permeabilizadas por 10 min com saponina 0,2% e 10% FCS (Thermo Fisher Scientific) em PBS. As células foram coradas com anti-ACE2 (1: 750, Abcam) ou anti-TMPRSS2 (1: 250, Abcam) e anticorpo anti-α-tubulina (1: 500, Thermo Fisher Scientific; Extended Data Fig.1 ) diluído em PBS, saponina 0,2% e FCS 10% durante a noite a 4 ° C. Posteriormente, as células foram lavadas duas vezes com PBS e incubadas por 1 h em temperatura ambiente em PBS, 0,2% de saponina e 10% de FCS contendo anticorpo secundário anti-coelho marcado com Alexa Fluor-488, anticorpo secundário anti-rato marcado com Alexa Fluor-647 (1: 500; Thermo Fisher Scientific) e DAPI (1: 5.000; Thermo Fisher Scientific). As imagens foram adquiridas em um microscópio confocal invertido (Leica TCS SP5) usando uma lente 40 × (Leica HC PL APO CS2 40 × 1,30 OIL). As imagens dos canais azul (DAPI), verde (Alexa Fluor-488) e vermelho (Alexa Fluor-647) foram obtidas no modo sequencial usando configurações de excitação e emissão adequadas.
Resumo de relatórios
Mais informações sobre o desenho da pesquisa estão disponíveis no Nature Research Reporting Summary vinculado a este artigo.
Disponibilidade de dados
Os dados de RNA-seq foram depositados no Gene Expression Omnibus do National Center for Biotechnology Information sob o número de acesso da série GEO GSE159717. Dados adicionais que apóiam os achados do presente estudo estão disponíveis junto aos autores correspondentes, mediante solicitação. Os dados de origem são fornecidos com este documento.
Disponibilidade de código
Para processamento de dados brutos fora dos instrumentos, o código para dois programas personalizados com base nas ferramentas Picard (v.2.19.2) está disponível em https://github.com/DanieleBarreca/picard e https://broadinstitute.github.io/picard . Outros programas usados para análise de transcriptoma são descritos em Métodos .
Referências
- 1
Zhu, N. et al. Um novo coronavírus de pacientes com pneumonia na China, 2019. N. Engl. J. Med. 382 , 727-733 (2020).
- 2
Lai, C.-C., Ko, W.-C., Lee, P.-I., Jean, S.-S. & Hsueh, P.-R. Manifestações extra-respiratórias de COVID-19. Int. J. Antimicrob. Agents 56 , 106024 (2020).
- 3 –
Gupta, A. et al. Manifestações extrapulmonares de COVID-19. Nat. Med. 26 , 1017–1032 (2020).
- 4 –
Wichmann, D. et al. Achados de autópsia e tromboembolismo venoso em pacientes com COVID-19: um estudo de coorte prospectivo. Ann. Estagiário. Med. 173 , 268-277 (2020).
- 5
Xiao, F. et al. Evidência de infecção gastrointestinal por SARS-CoV-2. Gastroenterology 158 , 1831-1833 e1833 (2020).
- 6
Puelles, VG et al. Tropismo multiorgânico e renal de SARS-CoV-2. N. Engl. J. Med. 383 , 590–592 (2020).
- 7
Song, E. et al. Neuroinvasion of SARS-CoV-2 no cérebro humano e de camundongo. J. Exp. Med. 218 , e20202135 (2021).
- 8
Hoffmann, M. et al. A entrada da célula SARS-CoV-2 depende de ACE2 e TMPRSS2 e é bloqueada por um inibidor de protease clinicamente comprovado. Cell 181 , 271–280.e8 (2020).
- 9
Liu, F. et al. A expressão de ACE2 no pâncreas pode causar dano pancreático após infecção por SARS-CoV-2. Clin. Gastroenterol. Hepatol. 18 , 2128–2130.e2 (2020).
- 10
Taneera, J. et al. O perfil de expressão dos receptores hospedeiros SARS-CoV-2 em ilhotas pancreáticas humanas revelou a regulação positiva de ACE2 em doadores diabéticos. Biology 9 , 215 (2020).
- 11
Blodgett, DM et al. Novas observações de sequenciamento de RNA de última geração de subconjuntos de células de ilhotas fetais e adultas humanas altamente purificadas. Diabetes 64 , 3172–3181 (2015).
- 12
Fignani, D. et al. A enzima conversora de angiotensina I do receptor SARS-CoV-2 tipo 2 (ACE2) é expressa em células β pancreáticas humanas e na microvasculatura do pâncreas humano. Frente. Endocrinol. 11 , 596898 (2020).
- 13
Kusmartseva, I. et al. Expressão dos fatores de entrada da SARS-CoV-2 no pâncreas de doadores de órgãos normais e indivíduos com COVID-19. Cell Metab. 32 , 1041–1051.e6 (2020).
- 14
Yang, JK, Lin, SS, Ji, XJ & Guo, LM A ligação do coronavírus SARS ao seu receptor danifica as ilhotas e causa diabetes agudo. Acta Diabetol. 47 , 193–199 (2010).
- 15
Muniangi-Muhitu, H. et al. Covid-19 e diabetes: uma relação bidirecional complexa. Frente. Endocrinol. 11 , 758 (2020).
- 16
Rubino, F. et al. Novo diabetes em Covid-19. N. Engl. J. Med. 383 , 789-790 (2020).
- 17
Guan, WJ et al. Características clínicas da doença coronavírus 2019 na China. N. Engl. J. Med. 382 , 1708–1720 (2020).
- 18
Li, J. et al. A infecção por COVID-19 pode causar cetose e cetoacidose. Diabetes Obes. Metab. 22 , 1935–1941 (2020).
- 19
Chow, N. et al. Estimativas preliminares da prevalência de condições de saúde subjacentes selecionadas entre pacientes com doença coronavírus 2019 – Estados Unidos, 12 de fevereiro a 28 de março de 2020. MMWR Morb. Mortal. Wkly Rep. 69 , 382-386 (2020).
- 20
Holman, N. et al. Fatores de risco para mortalidade relacionada ao COVID-19 em pessoas com diabetes tipo 1 e tipo 2 na Inglaterra: um estudo de coorte de base populacional. Lancet Diabetes Endocrinol. 8 , 823-833 (2020).
- 21
Williamson, EJ et al. Fatores associados à morte relacionada a COVID-19 usando OpenSAFELY. Nature 584 , 430-436 (2020).
- 22
Zhu, L. et al. Associação de controle de glicose no sangue e resultados em pacientes com COVID-19 e diabetes tipo 2 pré-existente. Cell Metab. 31 , 1068–1077.e3 (2020).
- 23
Akarsu, C. et al. Associação entre pancreatite aguda e COVID-19: poderia a pancreatite ser a peça que faltava no quebra-cabeça sobre o aumento das taxas de mortalidade? J. Invest. Surg. https://doi.org/10.1080/08941939.2020.1833263 (2020).
- 24
Xue, T. et al. Níveis de glicose no sangue em idosos com diabetes tipo 2 durante o surto de COVID-19: um estudo retrospectivo em um único centro. Pré- impressão em medRxiv https://doi.org/10.1101/2020.03.31.20048579 (2020).
- 25
Goldman, N., Fink, D., Cai, J., Lee, YN & Davies, Z. Alta prevalência de cetoacidose diabética associada a COVID-19 em cuidados secundários no Reino Unido. Diabetes Res. Clin. Pr. 166 , 108291 (2020).
- 26
Chee, YJ, Ng, SJH & Yeoh, E. Cetoacidose diabética precipitada por Covid-19 em um paciente com diabetes mellitus diagnosticado recentemente. Diabetes Res. Clin. Pr. 164 , 108166 (2020).
- 27
Marchand, L., Pecquet, M. & Luyton, C. Início do diabetes tipo 1 desencadeado por COVID-19. Acta Diabetol. 57 , 1265–1266 (2020).
- 28
Hollstein, T. et al. Diabetes mellitus insulino-dependente com autoanticorpo negativo após infecção por SARS-CoV-2: relato de caso. Nat. Metab. 2 , 1021–1024 (2020).
- 29
Ebekozien, OA, Noor, N., Gallagher, MP & Alonso, GT diabetes tipo 1 e COVID-19: resultados preliminares de um estudo de vigilância multicêntrico no US Diabetes Care 43 , e83-e85 (2020).
- 30
Unsworth, R. et al. Novo diabetes tipo 1 em crianças durante COVID-19: descobertas regionais multicêntricas no Reino Unido Diabetes Care 43 , e170 – e171 (2020).
- 31
Chen, J., Wu, C., Wang, X., Yu, J. & Sun, Z. O impacto de COVID-19 na glicose no sangue: uma revisão sistemática e meta-análise. Frente. Endocrinol. 11 , 574541 (2020).
- 32
Tittel, SR et al. O bloqueio COVID-19 afetou a incidência de diabetes tipo 1 pediátrico na Alemanha? Diabetes Care 43 , e172 – e173 (2020).
- 33
DiMeglio, LA, Albanese-O’Neill, A., Munoz, CE & Maahs, DM COVID-19 e crianças com diabetes – atualizações, incógnitas e próximos passos: primeiro, não faça extrapolações. Diabetes Care 43 , 2631–2634 (2020).
- 34
Coate, KC et al. Os fatores de entrada nas células SARS-CoV-2 ACE2 e TMPRSS2 são expressos na microvasculatura e nos dutos do pâncreas humano, mas não são enriquecidos em células β. Cell Metab. 32 , 1028–1040.e4 (2020).
- 35
Classificação do Diabetes Mellitus (OMS, 2019); https://www.who.int/publications/i/item/classification-of-diabetes-mellitus
- 36
Pan, L. et al. Características clínicas de pacientes com COVID-19 com sintomas digestivos em Hubei, China: um estudo descritivo, transversal e multicêntrico. Sou. J. Gastroenterol. 115 , 766–773 (2020).
- 37
Hikmet, F. et al. O perfil de expressão de proteínas de ACE2 em tecidos humanos. Mol. Syst. Biol. 16 , e9610 (2020).
- 38
Lee, JJ et al. Abundância relativa de genes de entrada SARS-CoV-2 nos enterócitos do trato gastrointestinal inferior. Genes 11 , 645 (2020).
- 39
Nicin, L. et al. Expressão específica do tipo celular do receptor ACE2 putativo da SARS-CoV-2 em corações humanos. EUR. Heart J. 41 , 1804-1806 (2020).
- 40
Chen, Y. et al. A enzima conversora de angiotensina Rhesus 2 suporta a entrada de coronavírus da síndrome respiratória aguda grave em macacos chineses. Virology 381 , 89-97 (2008).
- 41
Marques, FZ et al. Caracterização molecular dos componentes do sistema renina-angiotensina em tecidos intrauterinos humanos e membranas fetais de parto vaginal e cesariana. Placenta 32 , 214–221 (2011).
- 42
Smits, SL et al. Síndrome respiratória aguda grave distinta, vias de lesão pulmonar aguda induzida por coronavírus em duas espécies diferentes de primatas não humanos. J. Virol. 85 , 4234–4245 (2011).
- 43
Esumi, M. et al. A serina protease transmembrana TMPRSS2 ativa a infecção pelo vírus da hepatite C. Hepatology 61 , 437–446 (2015).
- 44
Collin, J. et al. A co-expressão dos genes de entrada da SARS-CoV-2 no epitélio da conjuntiva, limbo e córnea humana adulta superficial sugere uma via adicional de entrada através da superfície ocular. Ocul. Surf . https://doi.org/10.1016/j.jtos.2020.05.013 (2020).
- 45
Ubuka, T., Moriya, S., Soga, T. & Parhar, I. Identificação de serina 2 de protease transmembrana e caixa A1 de forkhead como os genes responsivos ao bisfenol A em potencial no cérebro de rato macho neonatal. Frente. Endocrinol. 9 , 139 (2018).
- 46
Zmora, P. et al. Ortólogos de primatas não humanos de TMPRSS2 clivam e ativam a hemaglutinina do vírus influenza. PLoS ONE 12 , e0176597 (2017).
- 47
Sakaguchi, W. et al. Existência de moléculas de entrada de SARS-CoV-2 na cavidade oral. Int. J. Mol. Sci. 21 , 6000 (2020).
- 48
Tsonkova, VG et al. A linha de células EndoC-betaH1 é um modelo válido de células beta humanas e aplicável para rastreios para identificar novos candidatos a drogas. Mol. Metab. 8 , 144–157 (2018).
- 49.
Blume, C. et al. Uma nova isoforma de ACE2 é expressa em epitélios respiratórios nasais e brônquicos humanos e é regulada positivamente em resposta à infecção por vírus respiratório de RNA. Nat Genet. https://doi.org/10.1038/s41588-020-00759-x (2021).
- 50
Lucas, JM et al. A serina protease TMPRSS2 do tipo II regulada por andrógeno é diferencialmente expressa e mal localizada no adenocarcinoma da próstata. J. Pathol. 215 , 118-125 (2008).
- 51
Leung, JM et al. Expressão de ACE-2 no epitélio das pequenas vias aéreas de fumantes e pacientes com DPOC: implicações para COVID-19. EUR. Respir. J . 55 , https://doi.org/10.1183/13993003.00688-2020 (2020).
- 52
Wang, M. et al. O remdesivir e a cloroquina inibem efetivamente o novo coronavírus recém-surgido (2019-nCoV) in vitro. Cell Res . 30 , 269-271 (2020).
- 53
Holland, AM, Hale, MA, Kagami, H., Hammer, RE & MacDonald, RJ Controle experimental do desenvolvimento e manutenção do pâncreas. Proc. Natl Acad. Sci. USA 99 , 12236–12241 (2002).
- 54
Wu, KL et al. O fator nuclear 3beta dos hepatócitos está envolvido na transcrição específica das células beta pancreáticas do gene pdx-1. Mol. Célula. Biol. 17 , 6002–6013 (1997).
- 55
Sander, M. et al. O gene homeobox Nkx6.1 encontra-se a jusante de Nkx2.2 na principal via de formação de células beta no pâncreas. Development 127 , 5533–5540 (2000).
- 56
Brahim Belhaouari, D. et al. Os pontos fortes da microscopia eletrônica de varredura na decifração do ciclo infeccioso SARS-CoV-2. Frente. Microbiol. 11 , 2014 (2020).
- 57
Zhu, N. et al. Morfogênese e efeito citopático da infecção por SARS-CoV-2 em células epiteliais das vias aéreas humanas. Nat. Comum. 11 , 3910 (2020).
- 58
Lamers, MM et al. O SARS-CoV-2 infecta de forma produtiva os enterócitos intestinais humanos. Science 369 , eabc1669 (2020).
- 59.
Semplici, F. et al. A mutação humana dentro do domínio Per-Arnt-Sim (PAS) contendo a proteína quinase (PASK) causa hipersecreção de insulina basal. J. Biol. Chem. 286 , 44005–44014 (2011).
- 60
Zhang, DD et al. Per-Arnt-Sim quinase (PASK): um regulador emergente da glicose em mamíferos e do metabolismo lipídico. Nutrients 7 , 7437–7450 (2015).
- 61
Boonen, SE et al. Diabetes neonatal transitória, ZFP57 e hipometilação de múltiplos loci impressos: um acompanhamento detalhado. Diabetes Care 36 , 505-512 (2013).
- 62
Hoytema van Konijnenburg, EMM et al. Hiperinsulinismo em um paciente com transtorno do espectro de Zellweger e síndrome de deleção 16p11.2. Mol. Genet Metab. Rep. 23 , 100590 (2020).
- 63
Huang, C. et al. A sinaptotagmina 4 regula a maturação das células beta pancreáticas modulando a sensibilidade ao Ca 2+ das vesículas de secreção de insulina. Dev. Cell 45 , 347–361 e345 (2018).
- 64
Aljaibeji, H. et al. A expressão reduzida de PLCXD3 está associada à interrupção da detecção de glicose e da sinalização de insulina em células β pancreáticas. Frente. Endocrinol. 10 , 735 (2019).
- 65
Pae, EK & Kim, G. Produção de insulina prejudicada por hipóxia intermitente via homeostase de zinco prejudicada. PLoS ONE 9 , e90192 (2014).
- 66
Ackermann, AM, Wang, Z., Schug, J., Naji, A. & Kaestner, KH Integration of ATAC-seq e RNA-seq identifica células alfa humanas e genes de assinatura de células beta. Mol. Metab. 5 , 233–244 (2016).
- 67
De Beeck, AO & Eizirik, DL Infecções virais em diabetes mellitus tipo 1 – por que as células β? Nat. Rev. Endocrinol. 12 , 263-273 (2016).
- 68
Ferreira, RC et al. Uma assinatura transcricional de interferon tipo I precede a autoimunidade em crianças geneticamente em risco de diabetes tipo 1. Diabetes 63 , 2538–2550 (2014).
- 69
Reynier, F. et al. Assinatura de expressão de gene específica associada ao desenvolvimento de diabetes autoimune tipo I usando análise de microarray de sangue total. Genes Immun. 11 , 269-278 (2010).
- 70
Kruger, J. et al . Inibição de drogas da replicação de SARS-CoV-2 em organóides intestinais derivados de células-tronco pluripotentes humanas. Cell Mol. Gastroenterol. Hepatol . https://doi.org/10.1016/j.jcmgh.2020.11.003 (2020).
- 71
Nienhold, R. et al. Dois perfis imunopatológicos distintos em pulmões de autópsia de COVID-19. Nat. Comum. 11 , 5086 (2020).
- 72
Menter, T. et al. O exame pós-morte de pacientes com COVID-19 revela dano alveolar difuso com congestão capilar grave e achados variados nos pulmões e outros órgãos, sugerindo disfunção vascular. Histopathology 77 , 198-209 (2020).
- 73
Braun, F. et al. O tropismo renal da SARS-CoV-2 está associado à lesão renal aguda. Lancet 396 , 597-598 (2020).
- 74
Farkash, EA, Wilson, AM & Jentzen, JM Evidência ultrastructural para infecção renal direta com SARS-CoV-2. Geléia. Soc. Nephrol. 31 , 1683-1687 (2020).
- 75
Hanley, B. et al. Achados histopatológicos e tropismo viral em pacientes do Reino Unido com COVID-19 fatal grave: um estudo post-mortem. Lancet Microbe 1 , e245 – e253 (2020).
- 76
Khan, S. et al. O SARS-CoV-2 infecta o rim? Geléia. Soc. Nephrol. 31 , 2746-2748 (2020).
- 77
Kissling, S. et al. Glomerulopatia em colapso em um paciente COVID-19. Kidney Int. 98 , 228-231 (2020).
- 78
Kudose, S. et al. Achados de biópsia renal em pacientes com COVID-19. Geléia. Soc. Nephrol. 31 , 1959–1968 (2020).
- 79
Su, H. et al. Análise histopatológica renal de 26 achados pós-morte de pacientes com COVID-19 na China. Kidney Int. 98 , 219-227 (2020).
- 80
Hasan, NM et al. Associação genética do gene da neuropilina-1 com diabetes tipo 1 em crianças: expressão da neuropilina-1 em ilhotas pancreáticas. Diabetes Res. Clin. Pr. 87 , e29-e32 (2010).
- 81
Segerstolpe, A. et al. Perfil de transcriptoma de célula única de ilhotas pancreáticas humanas na saúde e diabetes tipo 2. Cell Metab. 24 , 593–607 (2016).
- 82
Wang, YJ et al. Transcriptômica unicelular do pâncreas endócrino humano. Diabetes 65 , 3028–3038 (2016).
- 83
Cantuti-Castelvetri, L. et al. A neuropilina-1 facilita a entrada na célula SARS-CoV-2 e a infectividade. Science 370 , 856–886 (2020).
- 84
Wei, C. et al. O receptor B scavenger de HDL tipo 1 facilita a entrada de SARS-CoV-2. Nat. Metab. 2 , 1391–1400 (2020).
- 85
Hoffmann, M., Kleine-Weber, H. & Pöhlmann, S. Um sítio de clivagem multibásico na proteína de pico de SARS-CoV-2 é essencial para a infecção de células pulmonares humanas. Mol. Cell 78 , 779-784.e775 (2020).
- 86
Millet, JK & Whittaker, GR Proteases de células hospedeiras: determinantes críticos do tropismo e patogênese do coronavírus. Virus Res. 202 , 120–134 (2015).
- 87
Yang, L. et al. Uma plataforma baseada em células-tronco humanas pluripotentes para estudar o tropismo do SARS-CoV-2 e modelar a infecção por vírus em células humanas e organóides. Cell Stem Cell 27 , 125-136.e7 (2020).
- 88
Hodik, M. et al. A infecção por enterovírus em ilhotas humanas de Langerhans afeta a função das células beta, resultando em ilhotas desintegradas, diminuição da secreção de insulina estimulada por glicose e perda da estrutura de Golgi. BMJ Open Diabetes Res. Cuidado 4 , e000179 (2016).
- 89
Oshima, M. et al. A infecção semelhante a vírus induz a desdiferenciação de células beta humanas. JCI Insight 3 , e97732 (2018).
- 90
Sachs, S. et al. A terapia farmacológica direcionada restaura a função das células β para a remissão do diabetes. Nat. Metab. 2 , 192–209 (2020).
- 91.
Bozzo, CP et al. As proteínas IFITM promovem a infecção por SARS-CoV-2 e são alvos para a inibição do vírus. Pré- impressão em bioRxiv https://doi.org/10.1101/2020.08.18.255935 (2020).
- 92
Han, Y. et al. Identificação de inibidores da SARS-CoV-2 usando organoides de pulmão e cólon. Nature 589 , 270–275 (2021).
- 93.
Morris, KV A improbabilidade do rápido desenvolvimento de uma vacina para SARS-CoV-2. Mol. Ther. 28 , 1548–1549 (2020).
- 94
Bermejo-Martin, JF et al. A viremia de ARN da SARS-CoV-2 está associada a uma resposta do hospedeiro semelhante à sepse e doença crítica em COVID-19. Pré- impressão em medRxiv https://doi.org/10.1101/2020.08.25.20154252 (2020).
- 95
Fairweather, D. & Rose, NR diabetes tipo 1: infecção por vírus ou doença auto-imune? Nat. Immunol. 3 , 338-340 (2002).
- 96.
Ruiz, PL et al. Influenza pandêmica e risco subsequente de diabetes tipo 1: um estudo de coorte nacional. Diabetologia 61 , 1996–2004 (2018).
- 97
Petzold, A., Solimena, M. & Knoch, KP Mecanismos de disfunção das células beta associadas à infecção viral. Curr. Diab. Rep. 15 , 73 (2015).
- 98
Schepis, T. et al. Detecção de RNA SARS-CoV2 em uma amostra de pseudocisto pancreático. Pancreatology 20 , 1011–1012 (2020).
- 99
Desai, N. et al. Heterogeneidade temporal e espacial da resposta do hospedeiro à infecção pulmonar por SARS-CoV-2. Nat. Comum. 11 , 6319 (2020).
- 100
Brook, OR et al. Viabilidade e segurança da autópsia minimamente invasiva guiada por ultrassom em pacientes com COVID-19. Abdom. Radiol. https://doi.org/10.1007/s00261-020-02753-7 (2020).
- 101
Massoth, LR et al. Comparação de técnicas de hibridização in situ de RNA e imunohistoquímica para a detecção e localização de SARS-CoV-2 em tecidos humanos. Sou. J. Surg. Pathol. 45 , 14–24 (2021).
- 102
Best Rocha, A. et al. Detecção de SARS-CoV-2 em seções de tecido fixadas em formalina e embebidas em parafina usando reagentes disponíveis comercialmente. Lab. Investir. 100 , 1485-1489 (2020).
- 103
Bradley, BT et al. Histopatologia e achados ultraestruturais de infecções fatais por COVID-19 no Estado de Washington: uma série de casos. Lancet 396 , 320-332 (2020).
- 104
Love, MI, Huber, W. & Anders, S. Estimativa moderada de mudança de dobra e dispersão para dados de RNA-seq com DESeq2. Genome Biol. 15 , 550 (2014).
- 105
Raudvere, U. et al. g: Profiler: um servidor web para análise de enriquecimento funcional e conversões de listas de genes (atualização de 2019). Nucleic Acids Res. 47 , W191 – w198 (2019).
- 106
Kuleshov, MV et al. Enrichr: uma atualização de 2016 do servidor da web de análise de enriquecimento de conjunto de genes abrangente. Nucleic Acids Res. 44 , W90 – W97 (2016).
- 107
Subramanian, A. et al. Análise de enriquecimento de conjunto de genes: uma abordagem baseada no conhecimento para interpretar perfis de expressão em todo o genoma. Proc. Natl Acad. Sci. USA 102 , 15545–15550 (2005).
- 108
Groß, R. et al. Detecção de SARS-CoV-2 em leite materno humano. Lancet 395 , 1757-1758 (2020).
- 109
Chu, DK et al. Diagnóstico molecular de um novo coronavírus (2019-nCoV) causando um surto de pneumonia. Clin. Chem. 66 , 549–555 (2020).
- 110
Reed, LJ & Muench, H. Um método simples de estimar pontos finais de cinquenta por cento. Sou. J. Epidemiol. 27 , 493–497 (1938).
- 111
Groß, R. et al. Fragmento C-terminal derivado da placenta de β-hemoglobina com atividade antibacteriana e antiviral combinada. Frente. Microbiol. 11 , 508 (2020).
- 112
Picelli, S. et al. RNA-seq de comprimento total de células únicas usando Smart-seq2. Nat. Protoc. 9 , 171-181 (2014).
- 113
Picard toolkit v.2.19.2 (Broad Institute, 2019); https://github.com/DanieleBarreca/picard/
- 114
Dobin, A. et al. STAR: alinhador RNA-seq universal ultrarrápido. Bioinformatics 29 , 15–21 (2013).
- 115
Lawrence, M. et al. Software para computação e anotação de faixas genômicas. PLoS Comput. Biol. 9 , e1003118 (2013).
- 116
Wickham, H. ggplot2: Elegant Graphics for Data Analysis (Springer, 2016).
- 117
Gu, Z., Eils, R. & Schlesner, M. mapas de calor complexos revelam padrões e correlações em dados genômicos multidimensionais. Bioinformatics 32 , 2847–2849 (2016).
- 118
Blighe, K. & Sharmila Lewis, M. EnhancedVolcano: plotagens de vulcão prontas para publicação com coloração e rotulagem aprimoradas. Biocondutor de dentro do R. R versão 1.8.0 (2020).
- 119
Kolde, R. pheatmap package v. 1.0.12 (CRAN, 2018).
- 120
Stephens, M. Taxas de descoberta falsas: um novo negócio. Biostatistics 18 , 275–294 (2017).
- 121
Ignatiadis, N., Klaus, B., Zaugg, JB & Huber, W. A ponderação da hipótese baseada em dados aumenta o poder de detecção em testes múltiplos em escala de genoma. Nat. Methods 13 , 577–580 (2016).
- 122
Chen, EY et al. Enrichr: ferramenta de análise de enriquecimento de lista de genes HTML5 interativa e colaborativa. BMC Bioinf. 14 , 128 (2013).
- 123
Liberzon, A. et al. A coleção de conjuntos de genes de referência do banco de dados de assinaturas moleculares (MSigDB). Cell Syst. 1 , 417–425 (2015).
- 124
Winkelmann, VE et al. A suprarregulação da expressão de P2X4 induzida por inflamação aumenta a secreção de mucina no epitélio das vias aéreas. Sou. J. Physiol. Lung Cell. Mol. Physiol. 316 , L58 – L70 (2019).
Reconhecimentos
Agradecemos A. Saed, R. Gowdru Bijegatte, K. Köhn, D. Krnavek, N. Schrott, R. Kunz e J. Nell por sua excelente assistência técnica. Agradecemos também, em particular, L. Labriola, bem como M. Melzer, J. Merkle e M. Hohwieler, por discussões úteis. Agradecemos J. Lyon, N. Smith e J. Manning Fox (Alberta Diabetes Institute IsletCore) por seu trabalho de isolamento de ilhotas humanas e organizações de aquisição de órgãos em todo o Canadá, em particular o programa Human Organ Procurement and Exchange (HOPE) em Edmonton e no Trillium Gift of Life Network (TGLN) em Ontário, por seu trabalho na obtenção de pancreata humana para pesquisa. Agradecemos a I. Wessbecher, coordenador do banco de tecidos DZIF em Heidelberg, por fornecer cortes de tecido pancreático. Agradecemos também a S. Brandl pela assistência na organização de dados clínicos. Agradecemos S. Schmidt por compartilhar conosco a radiografia torácica e a história clínica do paciente 1. HL agradece R. Scharfmann por compartilhar a linha de células EndoC-βH1. Além disso, agradecemos K. Sato e M. Volcic por fornecer pLV-EF1a-ACE2-IRES-puro humano e as células HEK293T que expressam ACE2. O financiamento principal foi fornecido pela Deutsche Forschungsgemeinschaft (DFG) por meio de ‘financiamento de foco em COVID-19’ DFG KL 2544 / 8-1-AO 673221 para AK e JM, bem como por meio de ‘Sachbeihilfe’ KL 2544 / 7-1, ‘Heisenberg-Programm’ KL 2544 / 6-1 e a Baden-Württemberg-Foundation ExPoChip para AK Este trabalho foi financiado por doações do MWK Baden-Württemberg (para JAM, TFEB, JS, FK, JM e AK), o BMBF (restringir SARS-CoV-2 a FK), o programa de pesquisa e inovação Horizonte 2020 da UE (Fight-nCoV, 101003555 para JM) e o DFG (concessão nos. SPP1923 para KMJS e FK, e CRC1279 para KMJS, SS, FK e JM). KMJS é apoiado pelo Ministério Federal de Educação e Pesquisa da Alemanha (BMBF Junior Research Group IMMUNOMOD, 01KI2014). AK, MW e TS são os principais investigadores do HEIST RTG financiado pela DFG (GRK 2254/1). O financiamento adicional veio do DFG (KL 2544 / 1-1 e 1-2 e 5-1), e do financiamento de Excelência Else-Kröner-Fresenius (para AK). RG, CC, JK, LK e TW fazem parte, e RG é financiado por, uma bolsa de estudos da International Graduate School in Molecular Medicine, Ulm. O JAM agradece à Baden-Württemberg Stiftung pelo apoio financeiro a este projeto de pesquisa Eliteprogramme for Postdocs. SH recebeu fundos de apoio do Bausteinprogramm e é bolsista do Hertha-Nathorff-Programm da Universidade de Ulm. TE agradece o financiamento da DFG (bolsas nos. 380319649 e 376202546). O IGC recebeu financiamento da Iniciativa de Excelência dos governos federal e estadual da Alemanha. Este trabalho também foi financiado em parte pelo Centro Alemão de Pesquisa sobre Diabetes (DZD eV) e pela Helmholtz Alliance ‘Aging and Metabolic Programming, AMPro’.